
Protein adalah penting untuk kehidupan dan memainkan peranan dalam hampir setiap proses biologi. Di satu pihak, mereka boleh menghantar isyarat antara neuron, mengenal pasti penceroboh mikroskopik, dan mengaktifkan tindak balas imun. Sebaliknya, protein telah dikaji secara meluas sebagai mediator terapeutik sebagai sebahagian daripada rawatan penyakit. Oleh itu, dengan menghasilkan struktur protein baharu yang boleh dilipat secara fizikal, pintu dibuka kepada cara baharu untuk mengeksploitasi laluan selular untuk merawat penyakit.
Dalam artikel ini, penyelidik dari Universiti Stanford, Microsoft Research dan institusi lain, yang diilhamkan oleh proses lipatan protein dalam vivo, memperkenalkan model resapan lipat (folding diffusion, FoldingDiff)), yang mereka bentuk struktur tulang belakang protein dengan mencerminkan proses lipatan semula jadi protein.

Secara khusus, mereka menerangkan struktur tulang belakang protein sebagai satu siri sudut berturut-turut untuk menangkap orientasi relatif sisa-sisa asid amino konstituen, yang The intrinsik anjakan dan invarian putaran perwakilan ini sangat mengurangkan keperluan untuk rangkaian setara yang kompleks.
Kajian ini melatih model probabilistik resapan ternyah berdasarkan tulang belakang pengubah dan menunjukkan bahawa model kami tanpa syarat boleh menjana struktur protein yang sangat realistik dengan kerumitan dan corak struktur yang serupa dengan protein semula jadi.
Sesetengah netizen berkata: Saya tertanya-tanya jika model ini akan membawa beberapa persaingan kepada AlphaFold.
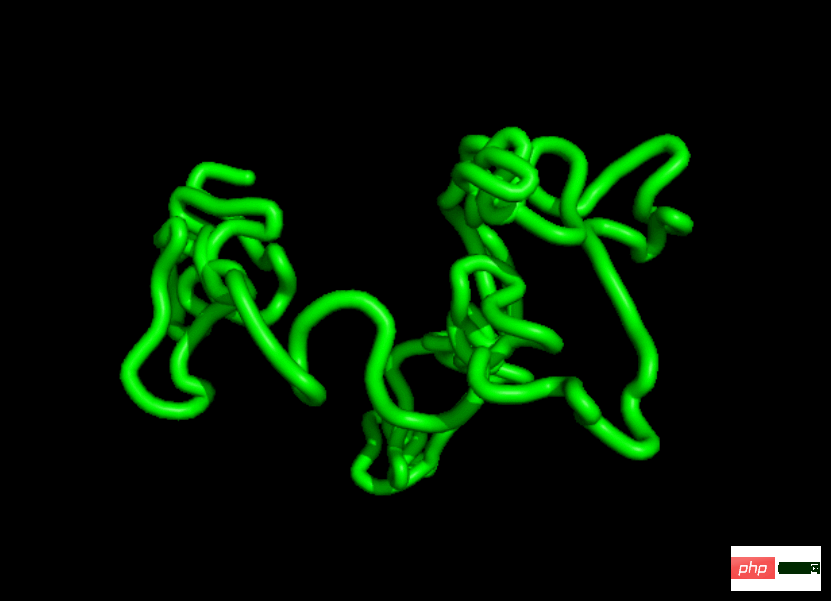
Kita boleh memahami protein sebagai rantaian sisa asid amino dengan panjang berubah-ubah spesies, berkongsi tulang belakang N-C_α-C tiga atom yang sama, tetapi dengan rantai sisi berbeza yang disambungkan kepada atom C_α (biasanya dilambangkan sebagai R, lihat Rajah 1).
Sisa-sisa ini berhimpun untuk membentuk rantai polimer yang terlipat menjadi struktur 3D, bentuk yang sebahagian besarnya menentukan fungsi protein. Struktur terlipat ini boleh diterangkan dalam empat peringkat:
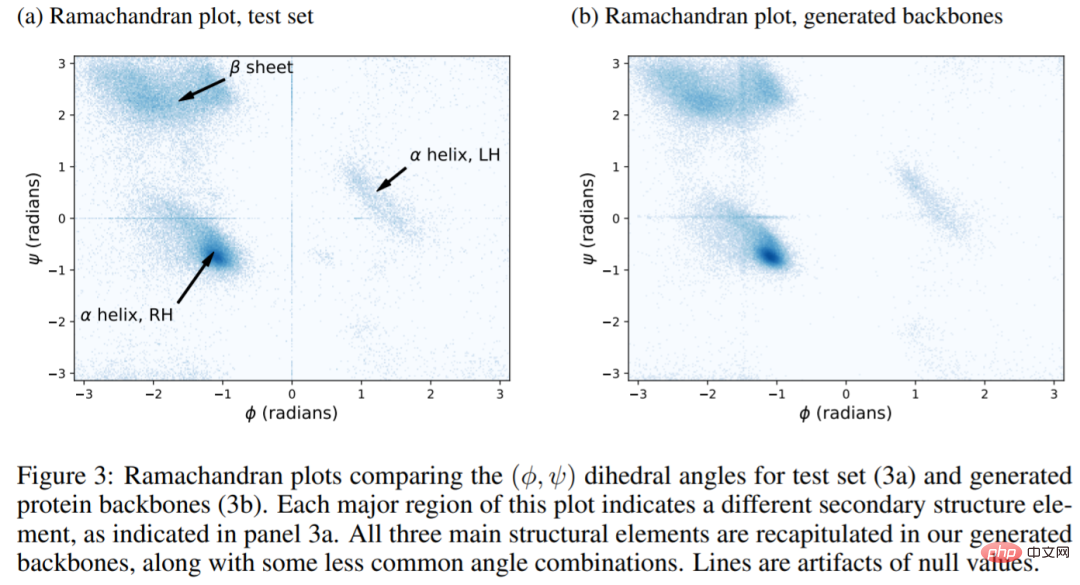
Gambar di bawah menunjukkan keputusan percubaan. Gambar rajah Ramachandran bagi struktur semula jadi (rajah a) mengandungi tiga kawasan yang sepadan dengan LH α-helix, RH α-helix, dan β-sheet. Ketiga-tiga rantau ini dihasilkan semula sepenuhnya dalam struktur yang dihasilkan di sini (Rajah 3b). Dengan kata lain, FoldingDiff mampu menjana elemen struktur sekunder dalam tulang belakang protein. Selain itu, eksperimen menunjukkan bahawa model FoldingDiff mengetahui dengan betul bahawa RH α-heliks adalah lebih biasa daripada LH α-heliks. Kerja sebelumnya menggunakan rangkaian setara tidak dapat membezakan antara kedua-dua jenis lingkaran ini.
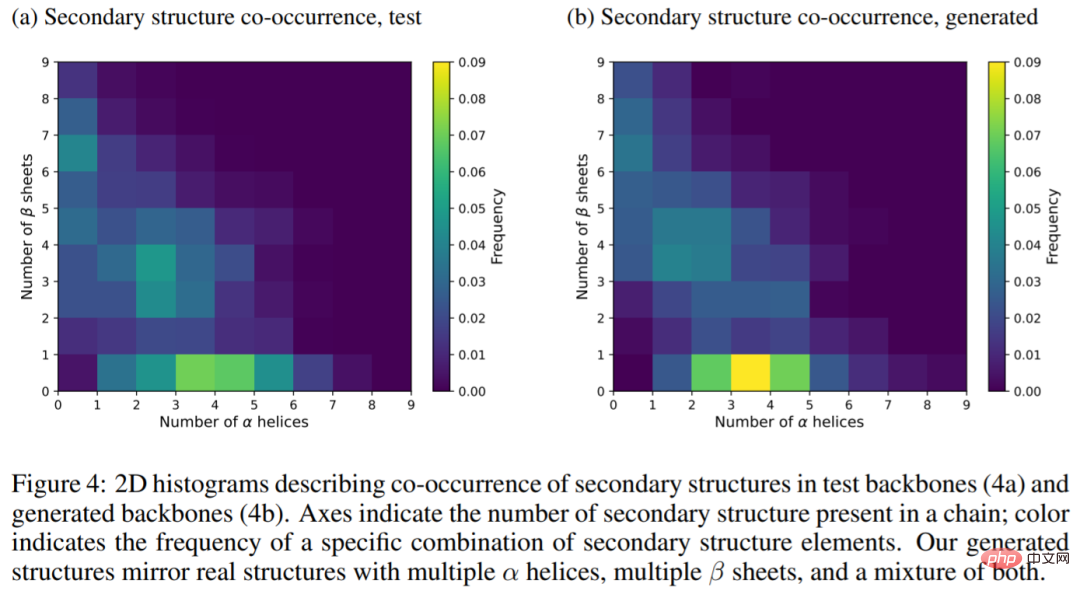
Gambar di bawah menunjukkan struktur sekunder dalam rantai utama ujian (4a) dan rantai utama yang dijana (4b) Dua -histogram dimensi, keputusan menunjukkan bahawa struktur yang dijana mencerminkan struktur sebenar protein, dengan berbilang α-heliks, berbilang helaian β, dan campuran kedua-duanya.
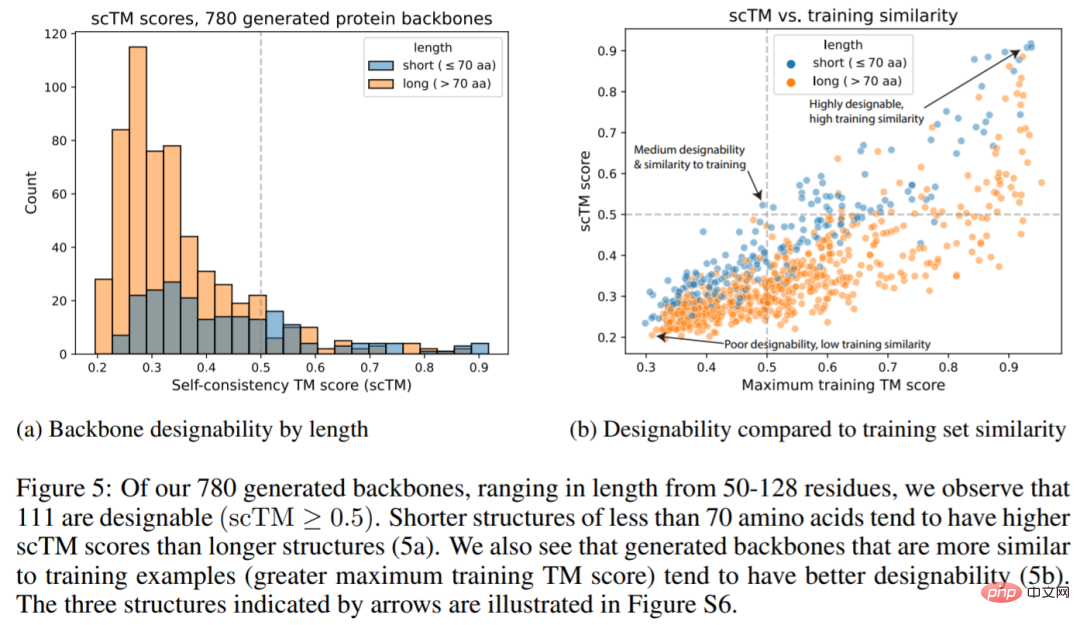
Rajah di bawah menunjukkan bahawa 111 daripada 780 struktur yang dijana (14.2%) boleh direka bentuk, dan skor scTMnya ≥0.5 (Rajah 5a) , iaitu lebih tinggi daripada nilai 11.8% yang dilaporkan oleh Trippe et al. Kami juga melihat bahawa rantai utama yang dihasilkan lebih serupa dengan contoh latihan dan cenderung mempunyai kebolehreka bentuk yang lebih baik (5b).
Untuk maklumat lanjut, sila baca kertas asal.
Atas ialah kandungan terperinci Stanford dan Microsoft bekerjasama untuk menggunakan model resapan untuk menjana struktur protein, yang telah menjadi sumber terbuka. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!




