AI在生物医学领域再次获得新进展。没错,这次还和蛋白质有关。
不同的是,过去的AI是发现蛋白质结构,这回开始自己设计和生成蛋白质结构了。如果说过去是「检察官」,现在说是进化成了「造物主」也不是不行。
参与本项研究的是Meta的AI研究机构中包括FAIR的蛋白质研究团队。作为在Facebook任职多年的首席AI科学家,Yann LeCun也是第一时间转发了这个自家团队的成果,并给予高度评价。

BioRxiv上的这两篇论文是Meta在蛋白质设计/生成方面的「惊人」的成果。该系统使用模拟退火算法来寻找一个氨基酸序列,该序列的折叠方式符合所需的形状或满足约束条件(如对称性)。
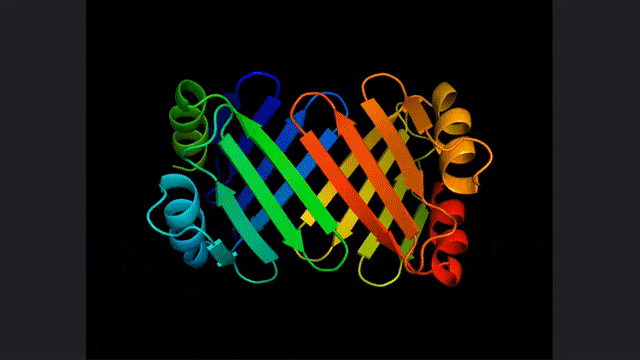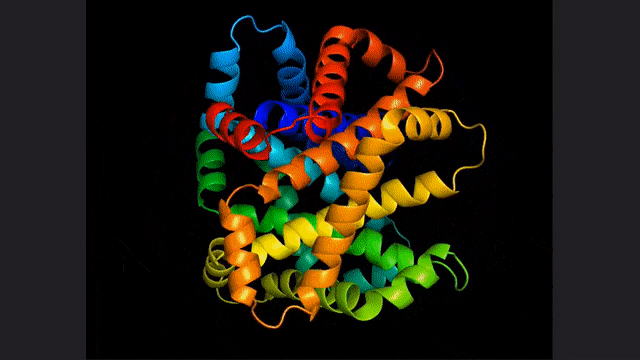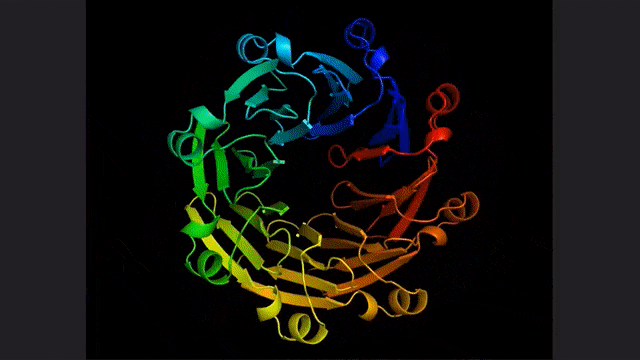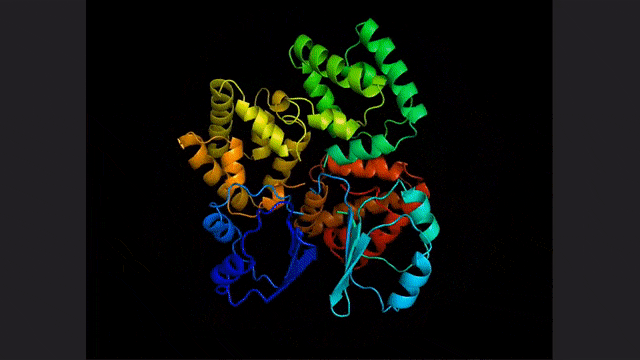
你猜的没错,这项研究和这两篇论文的基础,正是不久前由Meta提出的蛋白质预测和发现的大语言模型:ESM2。
这是一个150亿参数的大模型。随着模型从800万个参数扩展到1500万个参数,内部表征中出现的信息能够在原子分辨率下进行三维结构预测。
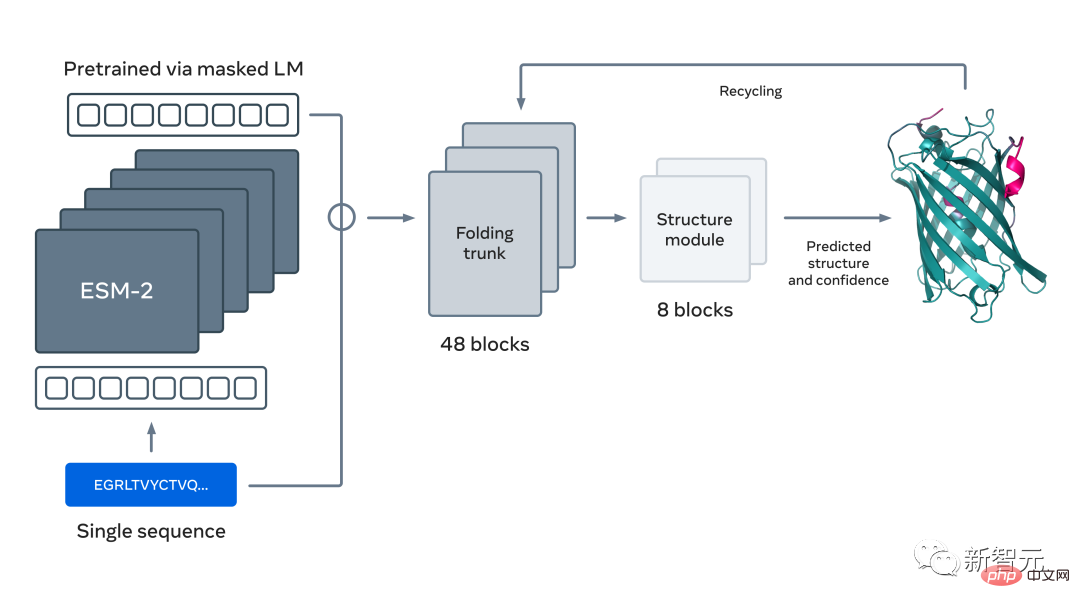
利用大型语言模型来学习进化模式,可以直接从蛋白质序列中端到端地生成准确的结构预测,在保持准确性的同时,预测速度比当前最先进的方法快60倍。
事实上,借助于这种新的结构预测能力,Meta在短短两周内用一个由大约2000个GPU组成的集群上,预测出了图谱中超过6亿个宏基因组蛋白质的序列。
两篇论文的通信作者,来自Meta AI的Alex Rives表示,ESM2语言模型展现出的通用性不仅超出了天然蛋白质的范围,而且还能够可编程地生成复杂和模块化的蛋白质结构。
工欲善其事,必先利其器。
为了让蛋白质设计和生成更有效率,研究人员在之前成果(主要是ESM2)的基础上,还专门开发了一种面向蛋白质设计的高级编程语言。
论文地址:https://www.biorxiv.org/content/10.1101/2022.12.21.521526v1
该研究的主要负责人之一,论文「A high-level programming language for generative protein design」的通讯作者Alex Rives在社交媒体上表示,这个成果,使得对具有复杂和模块化结构的大型蛋白质和复合物的生成进行编程成为可能。
论文作者之一、斯坦福大学的研究人员Brian Hie在推特上也对这篇文章的主要研究思路和成果做了自己的解释。
总体上讲,这篇文章描述了生成式机器学习如何实现由用于蛋白质设计的高级编程语言控制的复杂蛋白质的模块化设计。
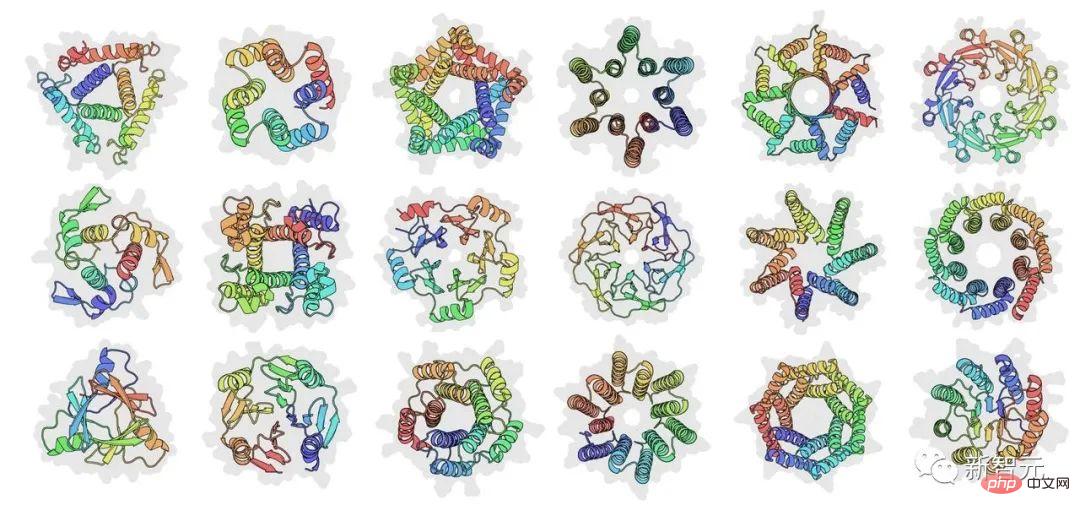
他表示,这篇文章的主要想法不是使用序列或结构的构建块,而是将模块化置于更高的抽象级别,并让黑盒优化生成特定设计。优化的每一步预测原子级结构。
与之前的蛋白质设计方法相比,这种新思路产生的方法可以让设计者指定任意的、不可微的约束,范围从指定原子级坐标到蛋白质的抽象设计方案,比如对称设计。
对于可编程性来说,约束条件是模块化的,这一点很重要。比如下图就是将同一约束分层应用于两个层次的对称性编程的情况。
这些约束也很容易重新组合。比如,可以把对原子坐标的约束和对对称性的约束结合起来。或者可以将不同形式的两级对称性结合起来,为一个不对称的复合结构体编程。
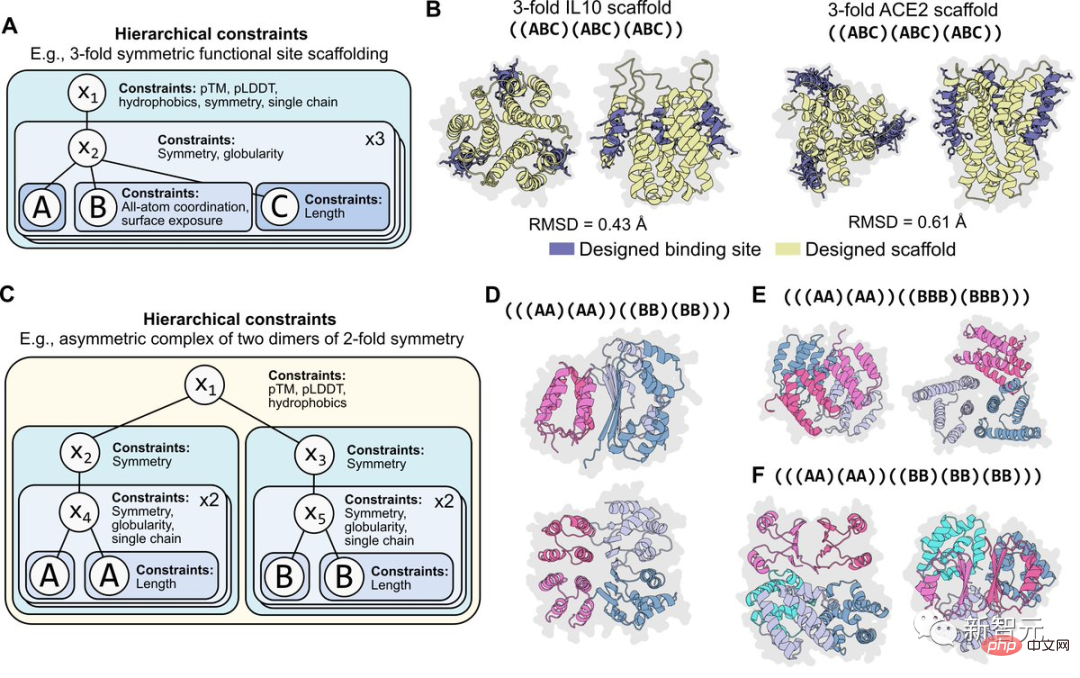
Brian Hie认为,这一成果是朝着更可控、更有规律、更有表现力的蛋白质设计迈出的一步。他也感谢了来自Meta AI和其他合作者的共同努力。
在论文中,研究人员认为,蛋白质设计将受益于一套基本的抽象概念所提供的规律性、简单性和可编程性,就像那些用于建筑、机器、电路和计算机软件工程的抽象概念一样。
但与这些人工创造物不同的是,蛋白质不能被分解成容易重组的部分,因为序列的局部结构与它的整体环境纠缠在一起。经典的从头开始的蛋白质设计试图确定一套基本的结构构件,然后将其组装成高阶结构。
同样,传统的蛋白质工程通常将天然蛋白质序列的片段或结构域重组为混合嵌合体。然而,现有的方法还不能实现真正可编程性所需的高组合复杂性。
本文展示了现代生成模型在新的组合复杂性水平上实现了模块化和可编程性的经典目标。把模块化和可编程性放在一个更高的抽象水平上,在这个水平上,生成式模型弥补了人类直觉和特定序列和结构的产生之间的差距。
在这种情况下,蛋白质设计者只需要重新组合高层次的指令,而获得满足这些指令的蛋白质的任务则放在生成模型上。
研究人员提出了一种用于生成性蛋白质设计的编程语言,允许设计者指定直观的、模块化的和分层次的程序。高层次的程序可以通过生成模型转化为低层次的序列和结构。这套方法利用了蛋白质语言模型的进展,可以学习结构信息和蛋白质的设计原则。
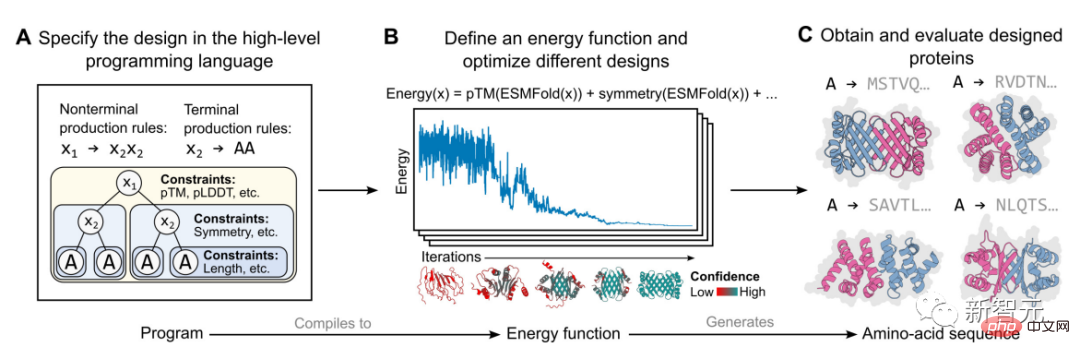
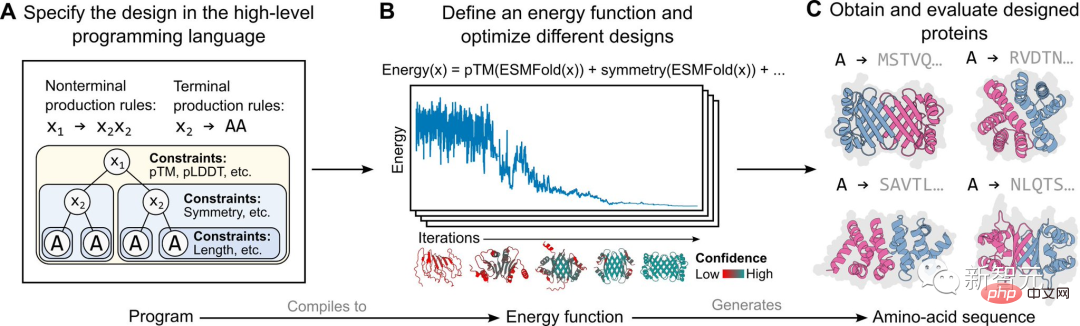
在这项研究中的具体实施基于一个基于能量的生成模型,如上图所示。
首先,一个蛋白质设计者指定了一个由一组分层组织的约束条件组成的高级程序(图A)。
然后,这个程序编译成一个能量函数,评估与约束条件的兼容性,这些约束条件可以是任意的和不可区分的(图B)。
通过将原子级结构预测(由语言模型启用)纳入能量函数来应用结构上的约束。这种方法能够生成广泛的复杂设计(图C)。
从无到有生成蛋白质序列
在论文「Language models generalize beyond natural proteins」中,来自MetaAI团队的作者Tom Sercu表示,这个工作主要完成了两项任务。

论文地址:https://www.biorxiv.org/content/10.1101/2022.12.21.521521v1
第一项是为给定的主链结构设计序列。使用语言模型,可以获得针对所有目标的成功设计,成功率达19/20,而没有语言模型参与的序列设计,成功率只有1/20。
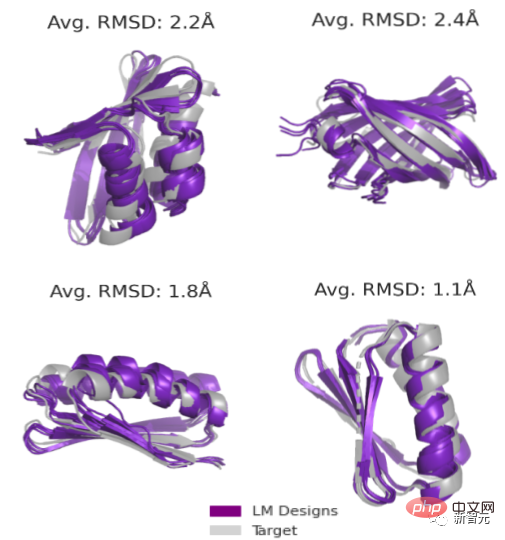
第二个任务是无约束生成。研究团队提出了一种从语言模型定义的能量景观中采样(序列、结构)对的新方法。

通过不同的拓扑结构进行采样,再次提高实验的成功率(达71/129或55%)。
为了证明预测的蛋白质结构超越了天然蛋白质的限制,研究团队在涵盖所有已知天然蛋白质的序列数据库中,对语言模型生成的蛋白质序列进行搜索。
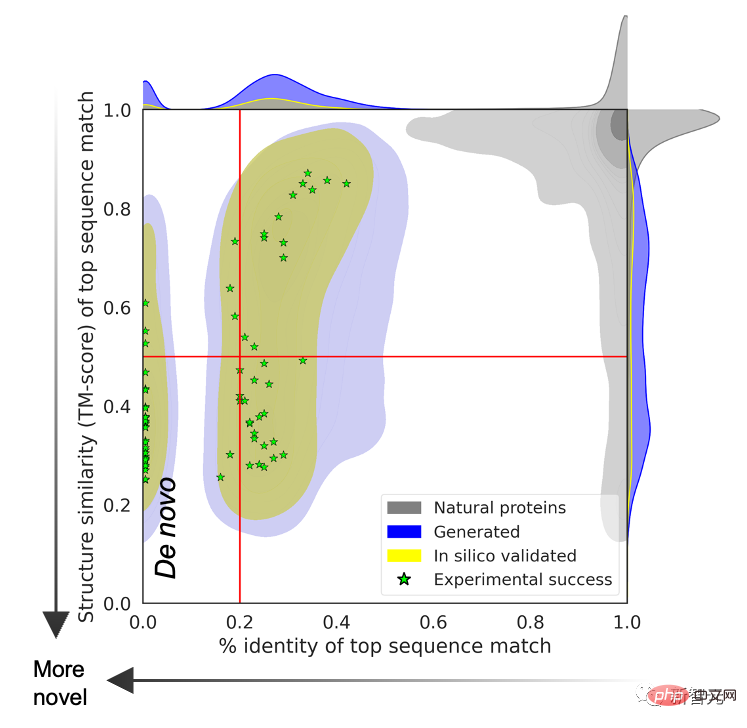
结果显示,二者并无匹配关系,自然序列和语言模型生成的预测结构不同。
Sercu表示,可以单独使用ESM2蛋白质语言模型对蛋白质结构进行设计。研究团队通过实验测试了228种蛋白质,成功率为67%!
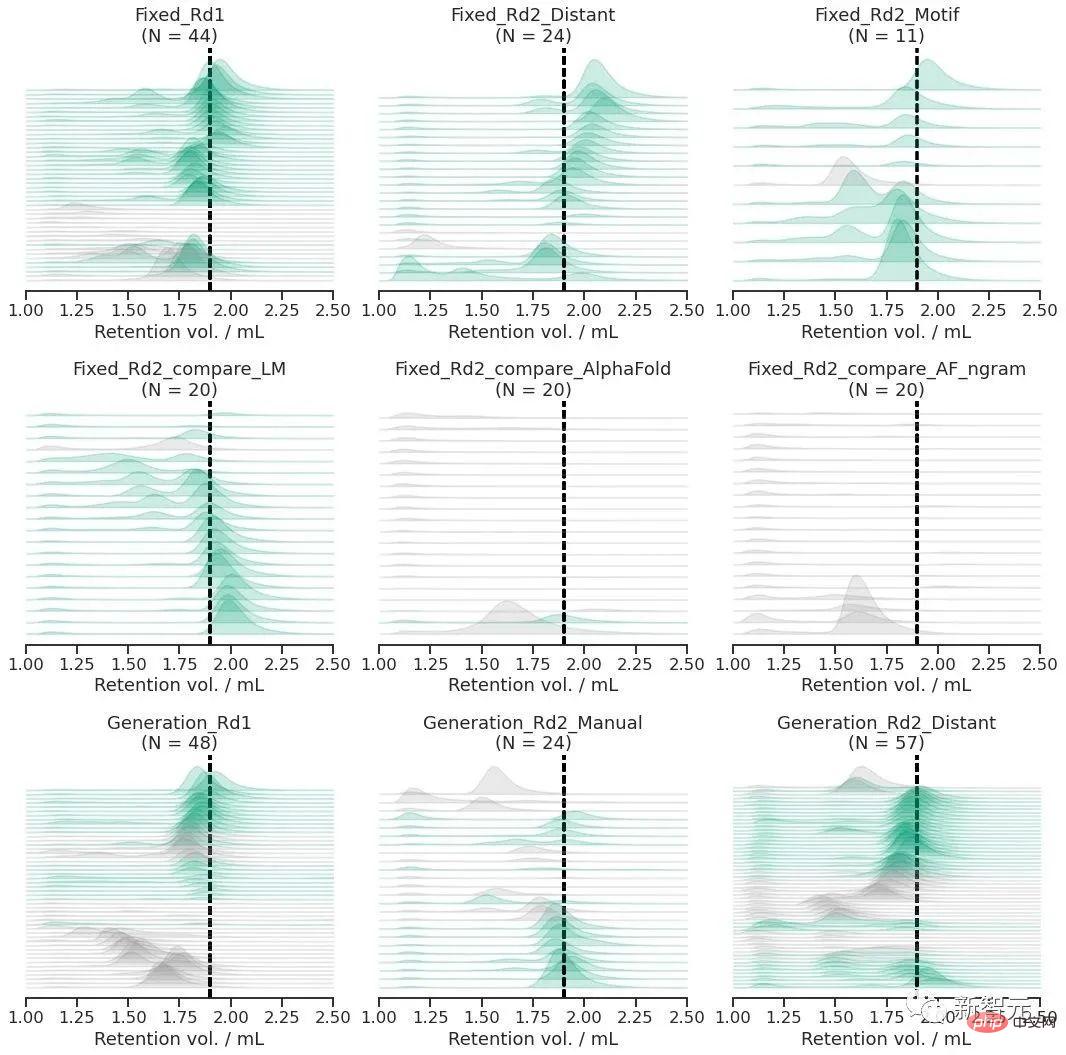
Sercu认为,仅在序列上训练的蛋白质语言模型可以学习连接序列和结构的深层模式,并且可以用于从头设计蛋白质,超出了自然探索的设计空间。
在论文中,Meta的研究人员表示,虽然只对语言模型在序列上训练,模型依然能够设计蛋白质的深层语法结构,突破天然蛋白质的局限。
如果用A图的方块表示所有蛋白质序列构成的空间,那么天然蛋白质序列是灰色部分,覆盖了其中一小部分。为了超越自然序列进行推广,语言模型需要访问底层设计模式。
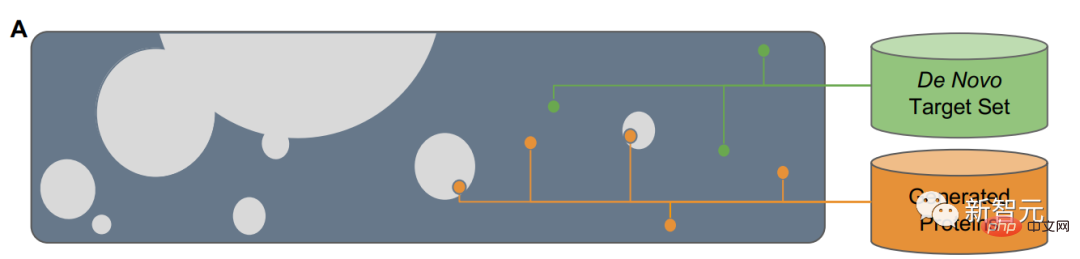
研究团队要做的是两件事:第一,从头设计蛋白质(de novo)主链;第二,根据主链,从无到有生成蛋白质序列。
研究团队使用掩码语言模型,对ESM2进行训练,训练内容包括进化过程中数百万种不同的天然蛋白质。
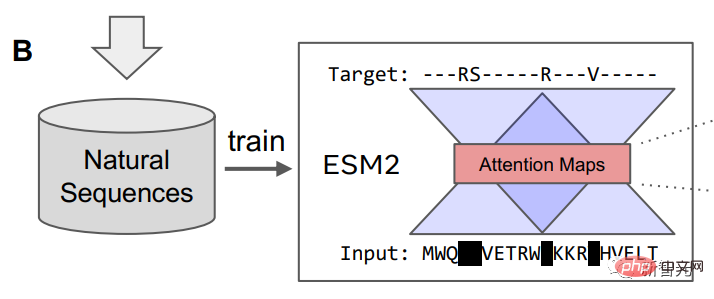
语言模型训练后,可以在模型的内部attention状态中识别有关蛋白质三级结构的信息。之后,研究人员通过线性投影,将蛋白质序列中一对位置的attention转换为残基间距离的分布。
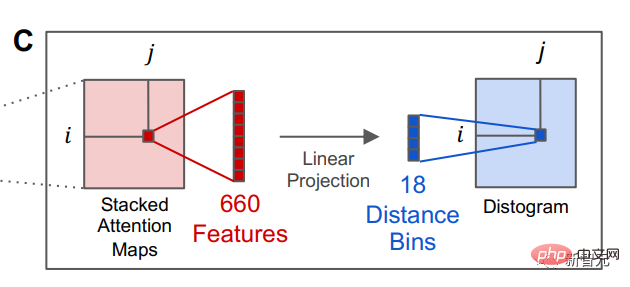
研究人员表示,语言模型预测蛋白质结构的能力,指出了构成天然蛋白质序列背后更深层次的结构序列,以及存在一个可由模型学习的深层语法的可能性。
结果表明,在进化过程中,大量蛋白质序列包含的生物结构和功能,揭示了蛋白质的设计构造。这种构造完全可以通过学习蛋白质序列的机器模型得以重现。
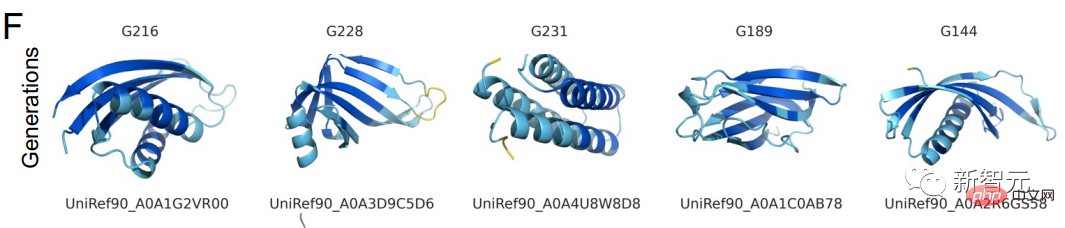
语言模型在6项实验中成功预测的蛋白质结构
跨蛋白质的深层语法的存在,解释了似乎相互矛盾的两组发现:对天然蛋白质的理解取决于训练数据;而语言模型又可以在已知的天然蛋白质家族之外进行预测和探索。
如果蛋白质语言模型的缩放定律继续有效,可以预料,AI语言模型的生成能力将不断提高。
研究团队表示,由于存在蛋白质结构的基础语法,机器模型将学习更加罕见的蛋白质构造,从而扩展模型的预测能力和探索空间。
一年前,DeepMind开源AlphaFold2连登Nature、Science,刷爆生物和AI学界。
一年后,人工智能预测模型如雨后春笋,频频填补蛋白质结构领域的空白。
如果说人类给予人工智能以生命,那么人工智能是否是人类补全生命奥秘的最后一块拼图呢?
以上是Meta让150亿参数语言模型学会从头设计「全新」蛋白质!LeCun:效果惊人的详细内容。更多信息请关注PHP中文网其他相关文章!




