
Model bahasa protein terbesar setakat ini telah dikeluarkan!
Setahun yang lalu, sumber terbuka DeepMind AlphaFold2 telah dilancarkan dalam Alam Semula Jadi dan Sains, mengatasi kalangan akademik biologi dan AI.
Setahun kemudian, Meta datang dengan ESMFold, yang merupakan susunan magnitud dengan lebih pantas.
Bukan sahaja ia pantas, model ini juga mempunyai 15 bilion parameter.
LeCun menulis tweet untuk memuji ini sebagai pencapaian baharu yang hebat oleh pasukan protein Meta-FAIR.

Pengarang bersama Zeming Lin mendedahkan bahawa model besar dengan 3 bilion parameter telah dilatih pada 256 GPU selama 3 minggu, manakala ESMfold mengambil masa 10 hari pada 128 GPU. Bagi versi parameter 15 bilion, ia masih tidak jelas.
Dia juga berkata bahawa kod itu pasti akan menjadi sumber terbuka nanti, jadi nantikan!
Hari ini, protagonis kami ialah ESMFold, model yang secara langsung meramalkan ketepatan tinggi, hujung ke hujung, struktur tahap atom daripada jujukan protein individu.

Alamat kertas: https://www.biorxiv.org/content/10.1101/2022.07.20.500902v1
Faedah 15 bilion parameter Tidak perlu katakan – model besar hari ini boleh dilatih untuk meramalkan struktur tiga dimensi protein dengan ketepatan bersaiz atom.
Dari segi ketepatan, ESMFold adalah serupa dengan AlphaFold2 dan RoseTTAFold.
Walau bagaimanapun, kelajuan inferens ESMFold adalah susunan magnitud lebih pantas daripada AlphaFold2!
Mungkin sukar untuk memahami perbandingan kelajuan antara ketiga-tiga dengan bercakap tentang susunan magnitud Lihat sahaja gambar di bawah.
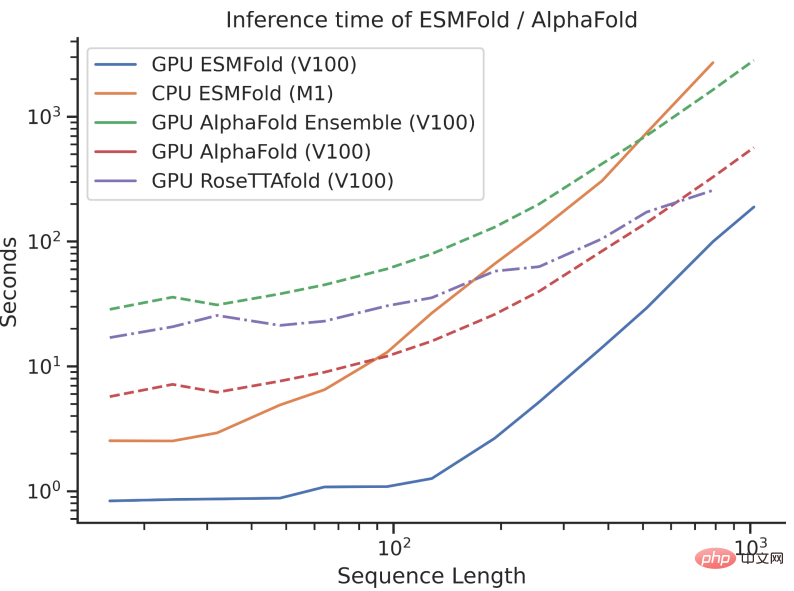
Walaupun AlphaFold2 dan RoseTTAFold telah mencapai kejayaan cemerlang dalam masalah ramalan struktur resolusi atom, mereka juga bergantung pada penggunaan penjajaran jujukan berbilang (MSA) dan serupa templat struktur protein untuk prestasi optimum.
Sebaliknya, dengan memanfaatkan perwakilan dalaman model bahasa, ESMFold boleh menjana ramalan struktur yang sepadan menggunakan hanya satu urutan sebagai input, sekali gus mempercepatkan ramalan struktur.
Para penyelidik mendapati bahawa ramalan ESMFold untuk jujukan kerumitan rendah adalah setanding dengan model terkini.
Selain itu, ketepatan ramalan struktur berkait rapat dengan kerumitan model bahasa, iaitu apabila model bahasa dapat memahami urutan dengan lebih baik, ia dapat memahami struktur dengan lebih baik.
Pada masa ini, terdapat berbilion jujukan protein struktur dan fungsi yang tidak diketahui, kebanyakannya berasal daripada jujukan metagenomik.
Menggunakan ESMFold, penyelidik boleh melipat sampel rawak 1 juta jujukan metagenomik dalam masa 6 jam sahaja.
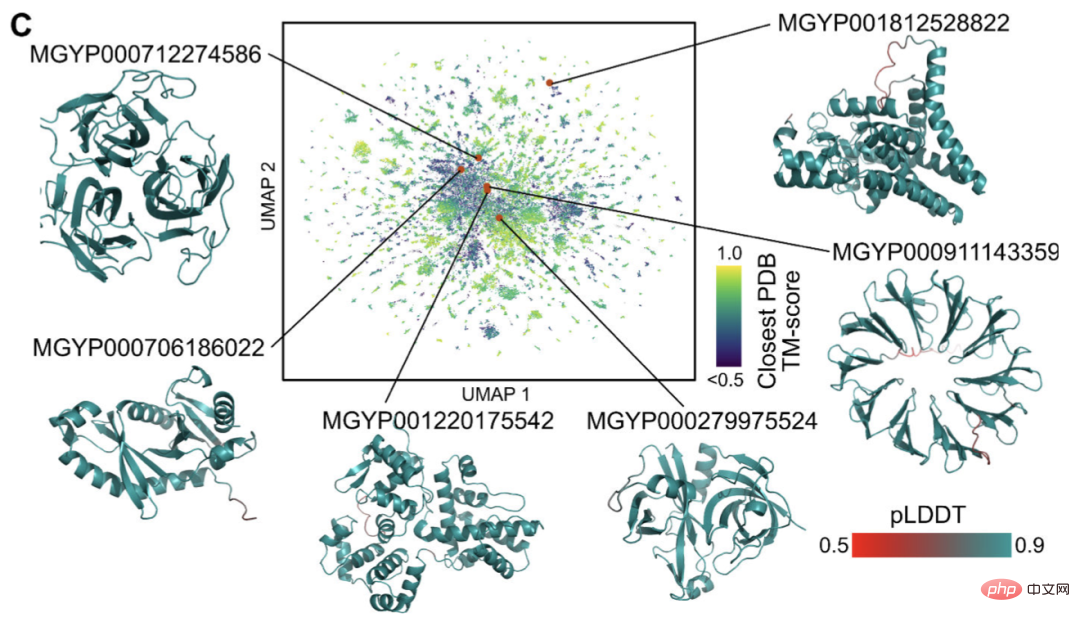
Sebahagian besar daripada ini mempunyai keyakinan yang tinggi dan tidak seperti mana-mana struktur yang diketahui (tidak direkodkan dalam pangkalan data).
Penyelidik percaya bahawa ESMFold boleh membantu memahami struktur protein yang di luar pemahaman semasa.
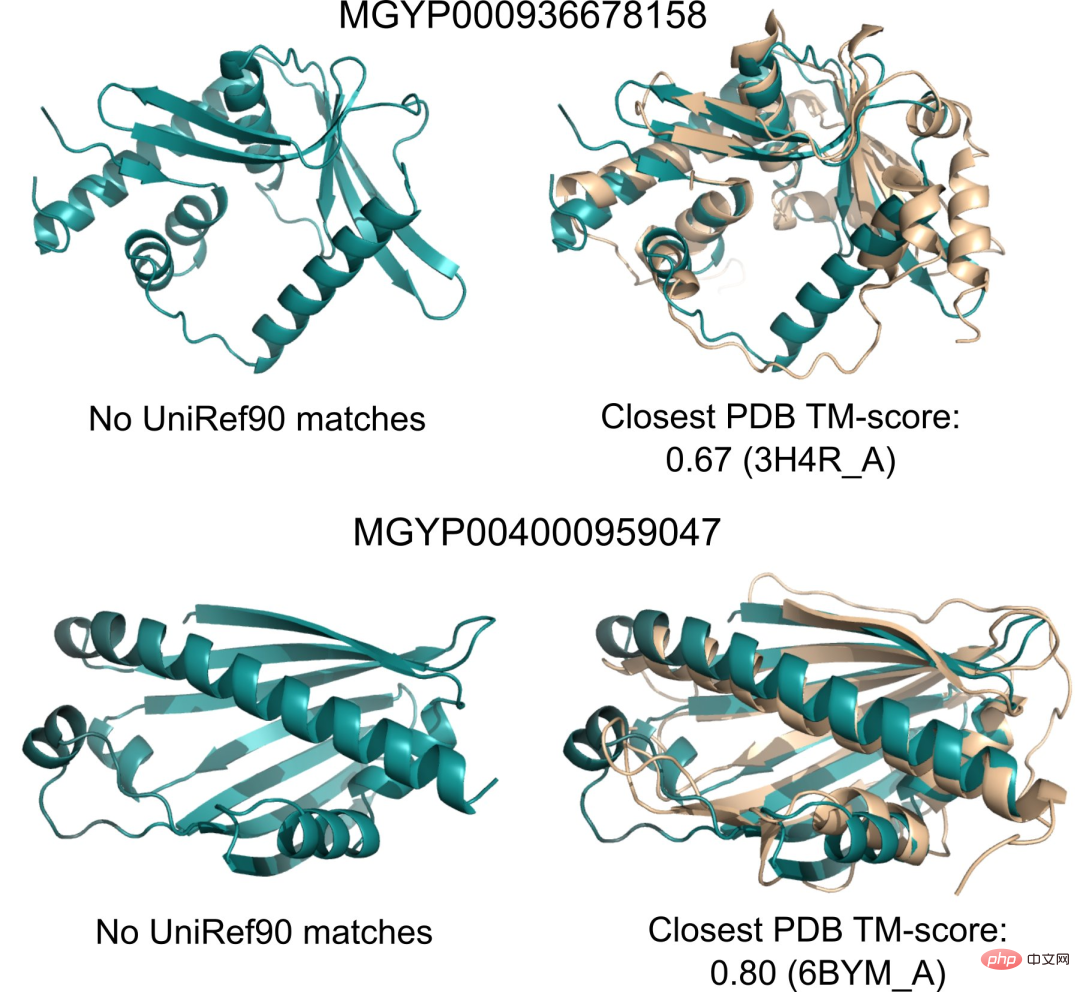
Di samping itu, kerana kelajuan ramalan ESMFold adalah susunan magnitud yang lebih pantas daripada model sedia ada, penyelidik boleh menggunakan ESMFold untuk membantu mengisi pangkalan data jujukan protein yang berkembang pesat dan kemajuan yang perlahan. Jurang antara struktur protein dan pangkalan data fungsi.
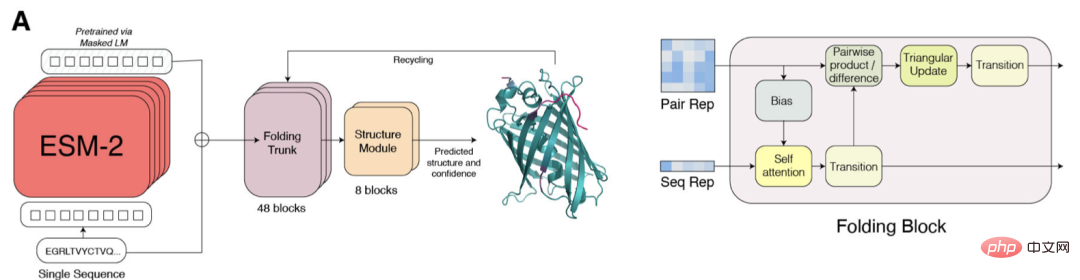
Seterusnya mari kita bincangkan tentang ESMFold baharu Meta secara terperinci.
ESM-2 ialah model bahasa berasaskan Transformer dan menggunakan mekanisme perhatian untuk mempelajari corak interaksi antara pasangan asid amino dalam jujukan input.
Berbanding dengan model generasi sebelumnya ESM-1b, Meta telah menambah baik struktur model dan parameter latihan, serta menambah sumber dan data pengkomputeran. Pada masa yang sama, penambahan benam kedudukan relatif membolehkan model digeneralisasikan kepada jujukan sebarang panjang.
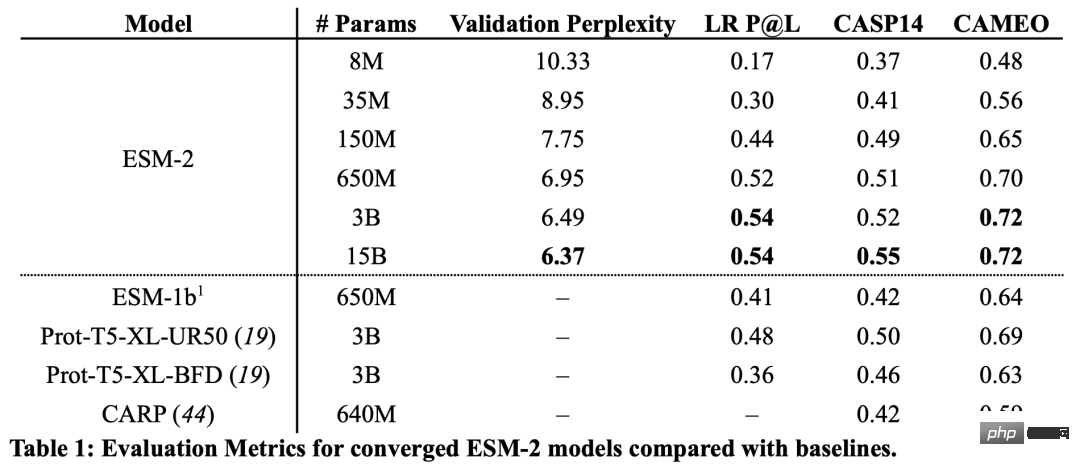
Daripada keputusan, model ESM-2 dengan 150 juta parameter berprestasi lebih baik daripada model ESM-1b dengan 650 juta parameter.
Selain itu, ESM-2 juga mengatasi model bahasa protein lain pada penanda aras ramalan struktur. Peningkatan prestasi ini adalah konsisten dengan corak yang telah ditetapkan dalam bidang pemodelan bahasa berskala besar.
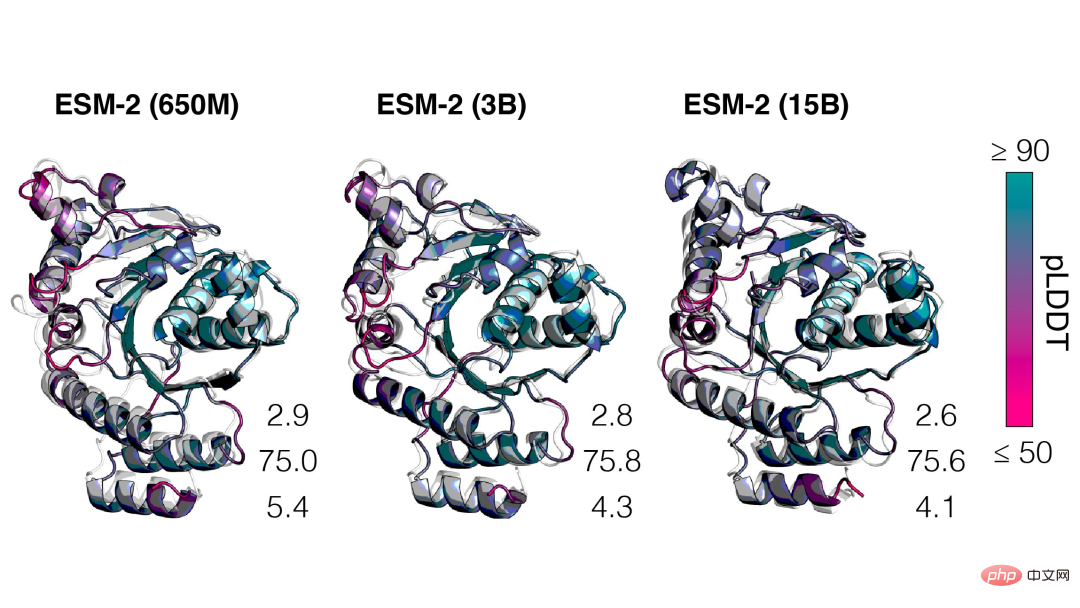
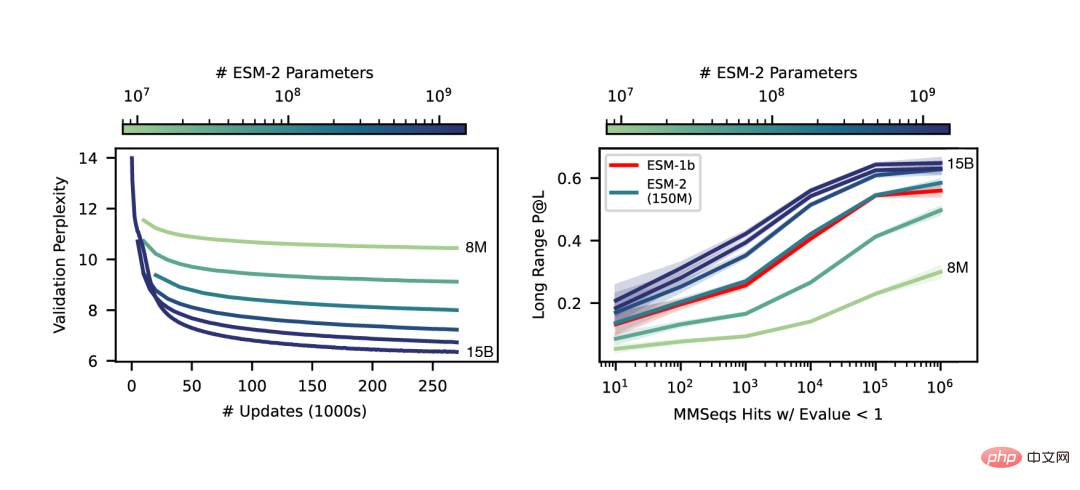
Apabila skala ESM-2 meningkat, peningkatan besar dalam ketepatan pemodelan bahasa dapat diperhatikan.
Perbezaan utama antara SMFold dan AlphaFold2 ialah ESMFold menggunakan perwakilan model bahasa, yang menghapuskan keperluan untuk Jujukan homologi eksplisit (dalam bentuk MSA) diperlukan sebagai input.
ESMFold memudahkan Evoformer dalam AlphaFold2 dengan menggantikan modul rangkaian yang mahal secara pengiraan yang mengendalikan MSA dengan modul Transformer yang mengendalikan jujukan. Penyederhanaan ini bermakna bahawa ESMFold adalah jauh lebih pantas daripada model berasaskan MSA.
Keluaran tulang belakang yang dilipat kemudiannya diproses oleh modul struktur, yang bertanggungjawab untuk mengeluarkan struktur tahap atom akhir dan keyakinan ramalan.
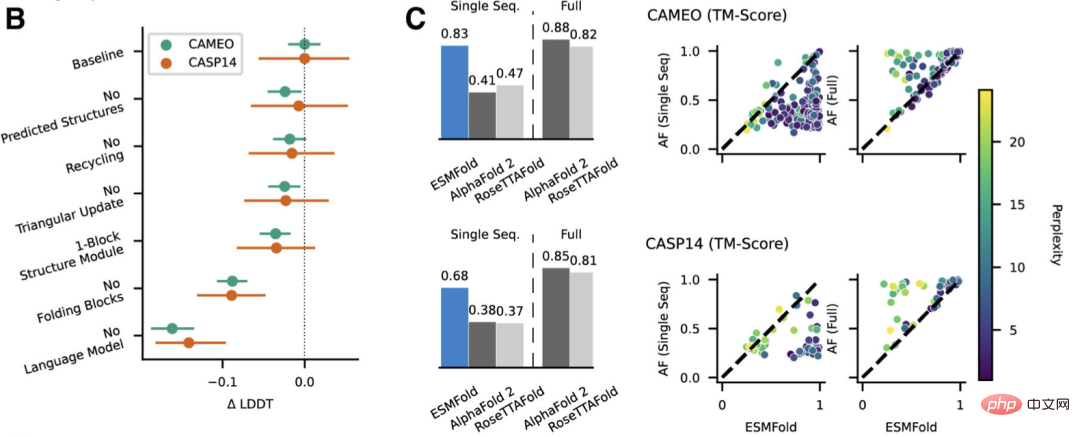
Penyelidik membandingkan ESMFold dengan AlphaFold2 dan RoseTTAFold pada set ujian CAMEO (April 2022 hingga Jun 2022) dan CASP14 (Mei 2020).
Apabila hanya satu jujukan diberikan sebagai input, ESMFold berprestasi lebih baik daripada Alphafold 2.
Apabila menggunakan saluran paip penuh, AlphaFold2 masing-masing mencapai 88.3 dan 84.7 pada CAMEO dan CASP14. ESMFold mencapai ketepatan yang setanding dengan RoseTTAfold pada CAMEO, dengan purata skor TM 82.0.
Para penyelidik mendapati bahawa model bahasa yang menyasarkan pembelajaran tanpa penyeliaan berprestasi baik pada yang besar. pangkalan data jujukan protein yang pelbagai secara evolusi, ia boleh meramalkan struktur protein dengan resolusi peringkat atom.
Dengan mengembangkan parameter model bahasa kepada 15B, kesan skala terhadap pembelajaran struktur protein boleh dikaji secara sistematik.
Kami melihat bahawa lengkung tak linear ramalan struktur protein ialah fungsi saiz model dan memerhatikan perkaitan yang kukuh antara sejauh mana model bahasa memahami jujukan dan ramalan struktur.
Model siri ESM-2 ialah model bahasa protein terbesar yang dilatih setakat ini, dengan hanya tertib magnitud parameter yang lebih sedikit daripada model teks terbesar yang dibangunkan baru-baru ini.
Selain itu, ESM-2 adalah peningkatan yang sangat besar berbanding model sebelumnya Walaupun pada parameter 150M, ESM-2 menangkap dengan lebih tepat daripada model bahasa generasi ESM-1 pada parameter 650M.
Penyelidik menyatakan bahawa pemacu terbesar prestasi ESMFold ialah model bahasa. Oleh kerana terdapat perkaitan yang kuat antara kebingungan model bahasa dan ketepatan ramalan struktur, mereka mendapati bahawa apabila ESM-2 dapat memahami urutan protein dengan lebih baik, ia boleh mencapai ramalan yang setanding dengan model terkini yang terkini.
ESMFold telah memperoleh ramalan struktur peleraian atom yang tepat, dan masa inferens adalah susunan magnitud lebih cepat daripada AlphaFold2.
Dalam amalan, kelebihan kelajuan adalah lebih hebat. Kerana ESMFold tidak perlu mencari jujukan berkaitan evolusi untuk membina MSA.
Walaupun terdapat cara yang lebih pantas untuk mengurangkan masa carian, ia mungkin masih sangat lama tidak kira berapa banyak anda mengurangkannya.
Faedah yang dibawa oleh masa inferens yang dipendekkan sangat jelas - peningkatan kelajuan akan memungkinkan untuk memetakan ruang struktur pangkalan data jujukan metagenomik yang besar.
Selain alat berasaskan struktur untuk mengenal pasti homologi dan pemuliharaan distal, ramalan struktur yang cepat dan tepat dengan ESMFold boleh memainkan peranan penting dalam analisis struktur dan fungsi bagi koleksi jujukan baharu yang besar.
Mendapatkan berjuta-juta struktur yang diramalkan dalam masa yang terhad akan membantu menemui pemahaman baharu tentang keluasan dan kepelbagaian protein semula jadi dan membolehkan penemuan struktur protein dan fungsi protein yang baharu sepenuhnya.
Pengarang bersama artikel ini ialah Zeming Lin daripada Meta AI.

Menurut halaman utama peribadinya, Zeming belajar untuk PhD di Universiti New York dan bekerja sebagai jurutera penyelidikan (melawat) di Meta AI, terutamanya bertanggungjawab untuk kerja infrastruktur bahagian belakang.
Dia belajar di University of Virginia untuk ijazah sarjana muda dan sarjana, di mana dia dan Yanjun Qi melakukan penyelidikan tentang aplikasi pembelajaran mesin, terutamanya dalam ramalan struktur protein.
Bidang yang diminati ialah pembelajaran mendalam, ramalan struktur dan biologi maklumat.
Atas ialah kandungan terperinci Lebih cepat daripada 0! Meta melancarkan model protein besar dengan 15 bilion parameter, menghancurkan AlphaFold2. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!
 Apakah perisian anti-virus?
Apakah perisian anti-virus?
 Platform mata wang digital domestik
Platform mata wang digital domestik
 Bagaimana untuk mengkonfigurasi pembolehubah persekitaran Tomcat
Bagaimana untuk mengkonfigurasi pembolehubah persekitaran Tomcat
 Apakah maksud c#?
Apakah maksud c#?
 Bagaimana untuk memulihkan fail yang dipadam secara kekal pada komputer
Bagaimana untuk memulihkan fail yang dipadam secara kekal pada komputer
 Bagaimana untuk membuka fail html pada telefon bimbit
Bagaimana untuk membuka fail html pada telefon bimbit
 Kaedah pemulihan pangkalan data Oracle
Kaedah pemulihan pangkalan data Oracle
 Bagaimana untuk menyelesaikan masalah semasa menghuraikan pakej
Bagaimana untuk menyelesaikan masalah semasa menghuraikan pakej




