

Pasukan penyelidik dari Universiti Tsinghua AIR, Universiti Peking dan Universiti Nanjing mencadangkan model ESM-AA. Model ini telah mencapai kemajuan penting dalam bidang pemodelan bahasa protein, menyediakan penyelesaian pemodelan bersatu yang menyepadukan maklumat berbilang skala.
Ia adalah model bahasa pra-latihan protein pertama yang boleh mengendalikan kedua-dua maklumat asid amino dan maklumat atom. Prestasi cemerlang model menunjukkan potensi besar pemodelan bersatu berbilang skala untuk mengatasi had sedia ada dan membuka kunci keupayaan baharu.
Sebagai model asas, ESM-AA telah mendapat perhatian dan perbincangan meluas daripada ramai sarjana (lihat tangkapan skrin di bawah), dan dianggap berpotensi untuk membangunkan model berasaskan ESM-AA yang boleh bersaing dengan AlphaFold3 dan RoseTTAFold All-Atom Ia membuka jalan baru untuk mengkaji interaksi antara struktur biologi yang berbeza. Kertas kerja semasa telah diterima oleh ICML 2024.
Protein ialah pelaksana utama pelbagai aktiviti kehidupan. Pemahaman mendalam tentang protein dan interaksinya dengan struktur biologi lain merupakan isu teras dalam sains biologi, yang mempunyai kepentingan praktikal yang signifikan untuk pemeriksaan dadah yang disasarkan, kejuruteraan enzim dan bidang lain.
Oleh itu, bagaimana untuk lebih memahami dan memodelkan protein telah menjadi tempat tumpuan penyelidikan dalam bidang AI4Science.
Dalam beberapa hari kebelakangan ini, institusi penyelidikan termaju utama, termasuk Deepmind dan Baker Group of the University of Washington, juga telah menjalankan penyelidikan mendalam tentang masalah pemodelan semua atom protein, dan kaedah yang dicadangkan termasuk AlphaFold 3, RoseTTAFold All-Atom, dsb. untuk protein dan aktiviti hidupan lain Model pemodelan skala semua atom bagi molekul berkaitan boleh mencapai ramalan tepat struktur protein, struktur molekul, struktur reseptor-ligan dan skala semua atom yang lain dengan ketepatan yang tinggi.
Walaupun model ini telah mencapai kemajuan yang ketara dalam pemodelan struktur pada skala semua atom, model bahasa protein arus perdana semasa masih tidak dapat mencapai pemahaman protein dan pembelajaran perwakilan pada skala semua atom.
Model pembelajaran perwakilan protein yang diwakili oleh ESM-2 menggunakan asid amino sebagai satu-satunya skala untuk membina model, yang sesuai untuk situasi yang memberi tumpuan kepada memproses protein adalah pendekatan yang munasabah.
Namun, kunci untuk memahami sepenuhnya sifat protein terletak pada menjelaskan interaksinya dengan struktur biologi lain seperti molekul kecil, DNA, RNA, dll.
Menghadapi permintaan ini, adalah perlu untuk menerangkan interaksi kompleks antara struktur yang berbeza, dan sukar bagi strategi pemodelan skala tunggal untuk menyediakan liputan komprehensif yang berkesan.
Untuk mengatasi kelemahan ini, model protein sedang menjalani inovasi mendalam ke arah model berskala. Sebagai contoh, model RoseTTAFold All-Atom yang diterbitkan dalam majalah Science pada awal Mei, sebagai produk susulan RoseTTAFold, memperkenalkan konsep berskala.
Model ini tidak terhad kepada ramalan struktur protein, tetapi juga meluas ke bidang penyelidikan yang lebih luas seperti dok protein dan molekul/asid nukleik, pengubahsuaian selepas terjemahan protein, dsb.
Pada masa yang sama, AlphaFold3 yang baru dikeluarkan oleh DeepMind juga menggunakan strategi pemodelan berbilang skala untuk menyokong ramalan struktur kompleks berbilang protein Prestasinya mengagumkan dan sudah pasti akan memberi impak besar pada bidang kecerdasan buatan dan biologi .
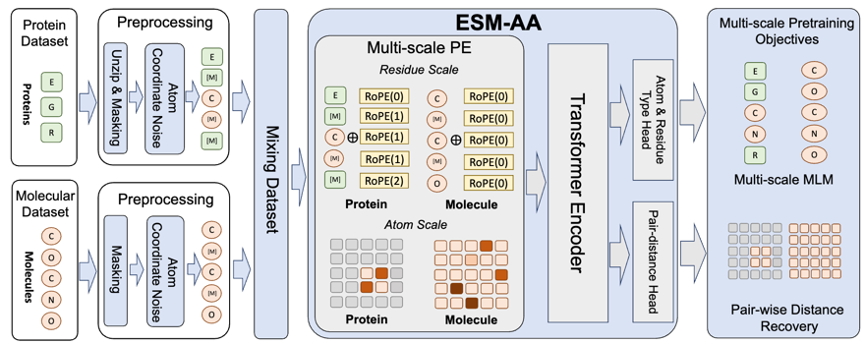
Penggunaan RoseTTAFold All-Atom dan Alpha-Fold3 yang berjaya pada konsep skala mengilhamkan Pemikiran penting ialah: bagaimana model bahasa protein sebagai model asas protein harus mengguna pakai teknologi pelbagai skala. Berdasarkan ini, pasukan mencadangkan model bahasa protein berskala ESM All-Atom (ESM-AA).
Secara ringkas, ESM-AA memperkenalkan konsep berskala berbilang dengan "nyahzip" beberapa asid amino ke dalam komposisi atom yang sepadan. Selepas itu, pra-latihan dilakukan dengan mencampurkan data protein dan data molekul, yang memberikan model keupayaan untuk mengendalikan struktur biologi skala yang berbeza pada masa yang sama.
Selain itu, untuk membantu model mempelajari maklumat skala atom berkualiti tinggi dengan lebih baik, ESM-AA juga akan menggunakan data struktur molekul skala atom untuk latihan. Selain itu, dengan memperkenalkan mekanisme pengekodan kedudukan berskala yang ditunjukkan dalam Rajah 2, model ESM-AA boleh membezakan maklumat dengan baik pada skala yang berbeza, memastikan model dapat memahami maklumat kedudukan dan struktur dengan tepat pada tahap sisa dan tahap atom.
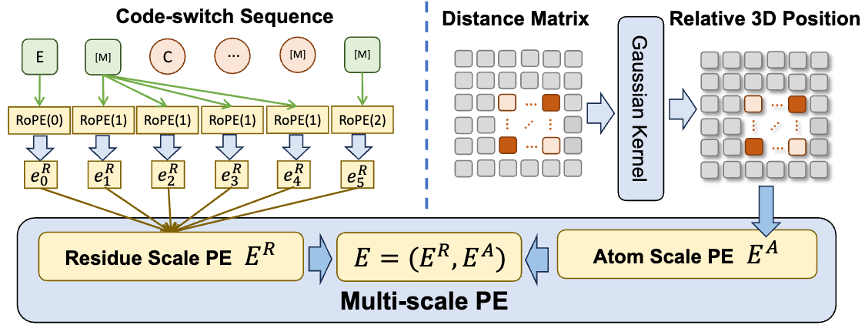
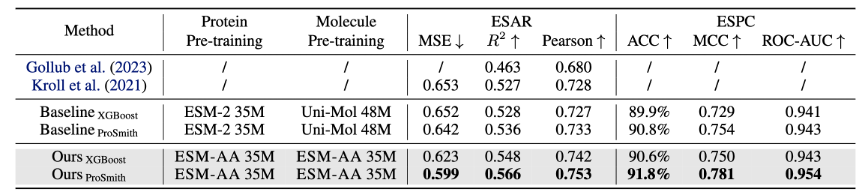
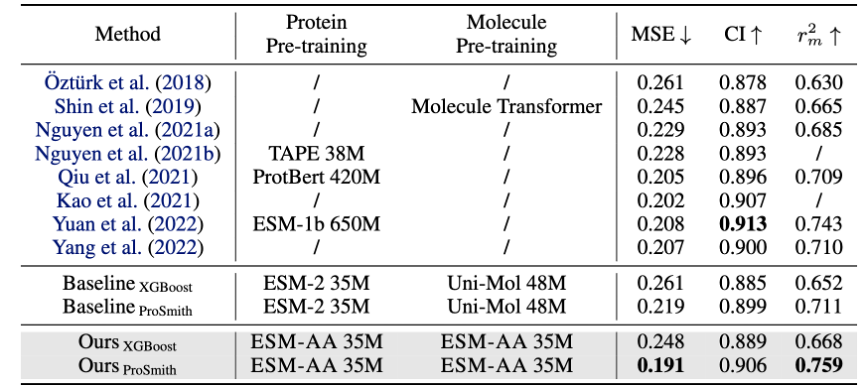
Untuk membantu model mempelajari maklumat berbilang skala, pasukan mereka bentuk pelbagai objektif pra-latihan untuk model ESM-AA. Objektif pra-latihan berbilang skala ESM-AA termasuk pemodelan bahasa bertopeng (MLM) dan pemulihan jarak berpasangan (PDR). Seperti yang ditunjukkan dalam Rajah 3(a), MLM memerlukan model untuk membuat ramalan berdasarkan konteks sekeliling dengan menutup asid amino dan atom Tugas latihan ini boleh dilakukan pada kedua-dua asid amino dan skala atom. PDR memerlukan model untuk meramalkan jarak Euclidean dengan tepat antara atom yang berbeza untuk melatih model memahami maklumat struktur peringkat atom (seperti ditunjukkan dalam Rajah 3(b)). . -tugas regresi pertalian bahan substrat (hasil ditunjukkan dalam Rajah 4), tugas pengelasan pasangan enzim-substrat (keputusan ditunjukkan dalam Rajah 4), dan tugas regresi pertalian sasaran dadah (keputusan ditunjukkan dalam Rajah 5).
Hasilnya menunjukkan bahawa ESM-AA mengatasi model sebelumnya dalam tugasan ini, menunjukkan bahawa ia menyedari sepenuhnya potensi model bahasa pra-latihan protein pada asid amino dan skala atom. 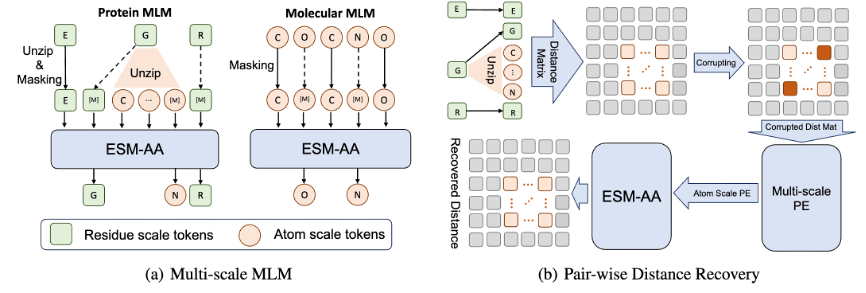
 Ini menunjukkan bahawa ESM-AA tidak mengorbankan keupayaannya untuk memahami protein dalam proses memperoleh pengetahuan molekul yang berkuasa Ia juga menggambarkan bahawa model ESM-AA berjaya menggunakan semula pengetahuan model ESM-2 tanpa perlu bermula dari. pembangunan, mengurangkan kos latihan model dengan ketara.
Ini menunjukkan bahawa ESM-AA tidak mengorbankan keupayaannya untuk memahami protein dalam proses memperoleh pengetahuan molekul yang berkuasa Ia juga menggambarkan bahawa model ESM-AA berjaya menggunakan semula pengetahuan model ESM-2 tanpa perlu bermula dari. pembangunan, mengurangkan kos latihan model dengan ketara.
Rajah 6: Analisis visual perwakilan protein/molekul
ESM-AA yang dibangunkan oleh pasukan Tsinghua AIR ialah model bahasa pemproses pra-latihan protein pertama yang mengintegrasikan asid amino dan maklumat atom. Model ini menunjukkan prestasi yang teguh dan cemerlang dengan menyepadukan maklumat berbilang skala, menyediakan cara baharu untuk menyelesaikan masalah interaksi antara struktur biologi.
ESM-AA bukan sahaja menggalakkan pemahaman yang lebih mendalam tentang protein, tetapi juga berfungsi dengan baik dalam pelbagai tugas biomolekul, membuktikan bahawa ia boleh mengintegrasikan pengetahuan peringkat molekul dengan berkesan sambil mengekalkan keupayaan pemahaman protein, mengurangkan kos latihan model hala tuju baharu untuk penyelidikan biologi berbantukan AI.
Tajuk kertas: ESM All-Atom: 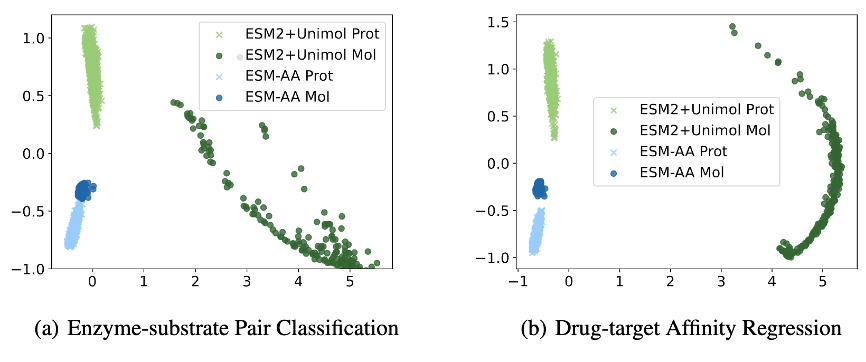 Model Bahasa Protein Berbilang Skala untuk Pemodelan Molekul Bersatu
Model Bahasa Protein Berbilang Skala untuk Pemodelan Molekul Bersatu
https://github.com/zhengkangjie/ESM-AA pautan:
https://arxiv.org/abs/2403.12995
Atas ialah kandungan terperinci Tsinghua AIR dan lain-lain mencadangkan ESM-AA, model bahasa protein pertama daripada asid amino kepada skala atom. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!




