
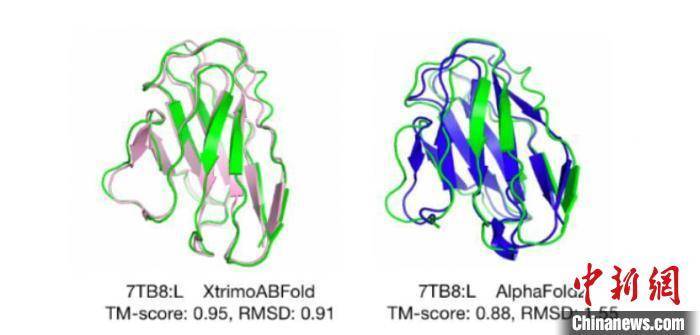
Baitu Biotechnology fournit une image comparant les performances de la prédiction de la structure des anticorps « AlphaFold2 » et « xTrimoABFold »
China News Service, Pékin, 12 juin Titre : Utiliser l'IA pour explorer la « clé » des anticorps et accélérer la recherche et le développement de médicaments - Entretien avec l'équipe de Baitu Biotechnology
Reporter Zhang Su
Imaginez, les protéines présentes dans le corps humain ont des « trous de serrure » de différentes formes. Si les chercheurs peuvent prédire avec précision les formes et concevoir les « clés » correspondantes, ils peuvent grandement améliorer l'efficacité du « déverrouillage », ce qui contribuera à accélérer le processus. de recherche et développement de médicaments plus avant-gardistes et d'autres projets liés aux sciences de la vie...
Le journaliste a appris de l'interview que l'équipe R&D de Baitu Biotech a réalisé une série de progrès positifs, notamment la sortie de la plateforme AIGP (AI Generated Protein) pilotée par de grands modèles des sciences de la vie.
Recherche et développement collaboratifs, idées et méthodes innovantes pour le développement de médicaments
Ces derniers temps, le laboratoire de Chen Zibo à l'Université de West Lake et Baitu Biotechnology ont mené conjointement un projet de recherche sur la conception de protéines à porte ET basé sur la plateforme AIGP. Cette initiative vise à concevoir des protéines artificielles « intelligentes » dotées de fonctions de porte logique moléculaire pour obtenir une réponse précise au signal et une régulation de systèmes vivants complexes et fournir des solutions innovantes pour le diagnostic et le traitement de nombreuses maladies.
Nous utilisons la plateforme AIGP pour concevoir et générer rapidement des protéines aux propriétés spécifiques. Cela fournit une base pour la conception et l’application de protéines à porte ET, et fournit également une nouvelle façon de penser et de nouvelles méthodes pour le développement de médicaments innovants. "Liu Wei, PDG de Baitu Biotech, a déclaré dans une interview.

La photo montre Liu Wei, PDG de Baitu Biotechnology. Photo fournie par Baitu Biotechnology
.Liu Wei a également cité l'exemple de sa coopération avec Logos Biotechnology pour explorer de nouvelles cibles tumorales, en utilisant la plateforme d'identification de cibles d'évasion immunitaire tumorale développée indépendamment (Tier-A) pour transformer rapidement les résultats innovants en un géant biologique doté d'un nouveau mécanisme d'action. "Il faut généralement plusieurs mois au laboratoire pour analyser la structure cristalline, mais la plateforme AIGP raccourcit ce processus à une ou deux semaines
."De l'avis de Song Le, directeur de la technologie et scientifique en chef de l'IA chez Baitu Biotechnology, la découverte de cibles, la synthèse et le criblage de composés sont des maillons clés dans la recherche et le développement de nouveaux médicaments. Les grands modèles d'IA devraient permettre d'obtenir une double amélioration de l'efficacité. et l'efficacité de ce processus, "Nous espérons concevoir des "clés" qui répondent plus rapidement à la précision du "trou de serrure" dans l'ordinateur, accélérant ainsi l'évolution des protéines conçues artificiellement, résolvant ainsi les problèmes de l'industrie des sciences de la vie."
Il est rapporté que Baitu Biotech divulguera bientôt une partie de l'interface utilisateur de la plateforme AIGP à des partenaires potentiels et à des chercheurs professionnels. Ils prévoient de lancer la version AIGP 2.0 d’ici un an pour fournir davantage de formation sur les modèles de mission et d’autres fonctionnalités.
Plus rapide et plus précis, favorisant l'itération du modèle et la mise en œuvre des applications
La plateforme AIGP avec ses nombreux points forts s'appuie sur le système de modélisation multimodale à grande échelle « xTrimo ».
Il est rapporté que « xTrimo » est composé d'un modèle pré-entraîné avec des centaines de milliards de paramètres et de plusieurs modèles de tâches en aval. Son modèle adopte la logique de conception d'une structure imbriquée à quatre couches. En avril de cette année, Baitu Biotech a publié des données montrant que le modèle de prédiction de la structure des anticorps « xTrimoABFold++ » présentait une précision et une efficacité supérieures. Par rapport à « AlphaFold2 », sa vitesse d'inférence est 540 fois plus rapide, sa vitesse d'exécution est 151 fois plus rapide et il peut prédire avec précision la structure atomique des anticorps en 3 secondes.
« L'exactitude ou la qualité des données aura un impact direct sur les performances du modèle. » a déclaré Song Le en expliquant les raisons de l'obtention de ce « bulletin ». L'équipe R&D a également mené un travail d'alignement détaillé sur les données publiques et complété les données de haute qualité avec des données de laboratoire internes pour relever efficacement les défis de l'exploration et de l'utilisation des données. La plate-forme de supercalcul biologique qu'ils ont créée auparavant peut obtenir dynamiquement des milliers à des dizaines de milliers de GPU et les ressources CPU correspondantes, ce qui est très puissant en termes de puissance de calcul.
Un autre sous-modèle du système « xTrimo », « xTrimoGene », et sa version « scFoundation » orientée vers des tâches plus en aval ont également reçu une large attention. Récemment, les résultats d'un article publié par Baitu Biotechnology et l'Université Tsinghua montrent que xTrimoGene et scFoundation surpassent les méthodes classiques dans six tâches de base et scénarios d'application en aval dans le domaine unicellulaire, notamment l'annotation du type de cellule, la prédiction des perturbations et la combinaison synergique de médicaments. Réalisation de SOTA industrie.
Notre objectif est de faire de notre mieux dans le travail de base et d'améliorer encore la précision et l'efficacité du modèle, notamment en augmentant la vitesse de prédiction de la structure des anticorps à quelques millisecondes. "Song Le a dit.
Dans le même temps, Liu Wei a déclaré qu'ils espéraient également ouvrir certaines capacités à l'industrie, recruter des partenaires plus écologiques, promouvoir l'itération et l'application rapides de grands modèles et former une boucle fermée de données de haute qualité.
Faire face à l'avenir, créer des protéines plus nombreuses et meilleures
Tout d’abord, les chercheurs scientifiques saisissent divers paramètres requis pour la protéine cible, et l’algorithme génère la protéine. Ensuite, la protéine est automatiquement imprimée et les chercheurs n'ont qu'à prendre la protéine produite automatiquement par l'algorithme pour une vérification scientifique plus approfondie... C'est la vision de Liu Wei pour la future « usine à protéines ».
Nous sommes impatients de pousser la plateforme AIGP vers un nouveau sommet grâce à nos efforts au cours des cinq prochaines années, en apportant de plus belles protéines au monde et en contribuant à créer une vie meilleure. ", a-t-il dit.
Baitu Biotech a lancé le « Excellent Developer Program », ouvert aux experts en biotechnologie de pointe, aux experts en développement de médicaments et aux équipes professionnelles cliniques, etc., et vise à cibler l'avenir. Song Le a expliqué que le plan fournira un financement de la recherche scientifique et un soutien technique aux projets de recherche médicale translationnelle de haut niveau. Il a déclaré que Baitu Biotechnology avait mis en place une équipe composée de talents aux antécédents multidisciplinaires pour garantir que les résultats de la recherche scientifique continuent d'être produits.
Il convient de noter qu'à mesure que le domaine d'application de la conception de protéines d'IA continue de s'étendre, la coopération menée par les parties concernées sur la base de la plate-forme AIGP ne se limite pas au sens étroit du développement de médicaments à base d'anticorps, mais s'étend également à une variété de médicaments à base de macromolécules. , la thérapie génique cellulaire, les outils des sciences de la vie, etc., et s'étend à la protection de l'environnement, au pétrole, à l'alimentation et à d'autres domaines, en utilisant la capacité de l'IA à générer des protéines innovantes pour résoudre des problèmes plus pratiques. Par exemple, les chercheurs étudient les protéases dans le but de trouver un moyen de décomposer efficacement les plastiques ou d’accélérer les préparations pour une production d’énergie spécifique.
Liu Wei a déclaré que tout en travaillant dur pour élargir les domaines cibles et les modèles commerciaux, ils maintiennent la même intention initiale, à savoir « utiliser de grands modèles d'IA, une technologie de bioinformatique de pointe, etc. pour modéliser des formes de vie complexes et concevoir de nouvelles protéines. « pour relever les défis ». Malgré l’énorme investissement, ils continueront à avancer fermement sur cette voie. (Fin)
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
 Utilisation de la fonction qsort
Utilisation de la fonction qsort
 utilisation de location.reload
utilisation de location.reload
 Que sont les bases de données non relationnelles ?
Que sont les bases de données non relationnelles ?
 Comment effacer le flotteur en CSS
Comment effacer le flotteur en CSS
 Quel est le problème avec le déclenchement de l'interrupteur pneumatique ?
Quel est le problème avec le déclenchement de l'interrupteur pneumatique ?
 Comment ouvrir les fichiers .dat
Comment ouvrir les fichiers .dat
 Comment acheter et vendre du Bitcoin sur Binance
Comment acheter et vendre du Bitcoin sur Binance
 Méthodes pour prévenir les attaques CC
Méthodes pour prévenir les attaques CC