
Kurs Dazwischenliegend 11245
Kurseinführung:《自学IT网Linux负载均衡视频教程》主要通过对web,lvs以及在nagin下对Linux进行脚本操作来实现Linux负载均衡。

Kurs Fortschrittlich 17581
Kurseinführung:《尚学堂MySQL视频教程》向大家介绍了如何从安装到使用MySQL数据库的这么一个过程,详细的介绍的每个环节的具体操作。

Kurs Fortschrittlich 11291
Kurseinführung:《兄弟连前端实例展示视频教程》向大家介绍了html5和css3技术的实例,让大家更加熟练掌握使用html5和css3.
Wie schalte ich die lästigen Tastenkombinationen in phpstudy aus?
2018-12-12 11:31:43 0 1 1201
In React js erfahren Sie, wie Sie mit useState den Schlüsselwert eines Objekts aktualisieren
constRow=[{name:"jad",link:"/jad",Kalorien:"52",Fett:"4",Kohlenhydrate:"5",Protein:"41"},{name:&q
2023-09-13 21:31:08 0 1 563
javascript - Handelt es sich wieder um ein Prototypproblem?
2017-05-19 10:30:56 0 3 539
2023-09-05 11:18:47 0 1 751

Kurseinführung:Herausgeber | Rettichhaut Um die Funktion von Proteinen zu verstehen und molekulare Therapien zu entwickeln, müssen die Zelltypen identifiziert werden, in denen das Protein wirkt, und die Wechselwirkungen zwischen Proteinen analysiert werden. Allerdings bleibt die Modellierung von Proteininteraktionen über biologische Kontexte hinweg eine Herausforderung für bestehende Algorithmen. In der neuesten Studie entwickelten Forscher der Harvard Medical School PINNACLE, eine geometrische Deep-Learning-Methode zur Generierung kontextbezogener Proteindarstellungen. PINNACLE nutzt Multiorgan-Einzelzellatlanten, um kontextualisierte Proteininteraktionsnetzwerke zu erlernen und generiert 394.760 Proteindarstellungen aus 156 Zelltypkontexten in 24 Geweben. Die Studie basiert auf dem Konzept „ContextualAImodelsforsingl“.
2024-07-26 Kommentar 0 1140

Kurseinführung:Editor |. KX Im Leben ist es einfach, eine Lampe einzuschalten oder die Beleuchtung anzupassen. Doch Systeme, die eine ähnliche Kontrolle biomolekularer Funktionen erreichen, sind komplex und wenig verstanden. In der Biologie werden Proteinfunktionen auf komplexe Weise ein- und ausgeschaltet, und die allosterische Regulierung ist einer der wichtigen biologischen Regulierungsmechanismen, die für einen gesunden Stoffwechsel und die Zellsignalisierung unerlässlich sind. Aber die Herstellung von Allosterie in synthetischen Proteinsystemen war schon immer mit großen Herausforderungen verbunden. Kürzlich hat das Team von David Baker an der University of Washington ein Protein entwickelt, das durch allosterische Kontrolle zuverlässig und genau zwischen Auf- und Abbau wechseln kann. Die Forscher nutzten KI, um neue Proteine zu entwerfen, die in der Natur nicht vorkommen, und entwarfen eine Vielzahl dynamischer Proteinanordnungen. David Baker sagte:
2024-08-20 Kommentar 0 676
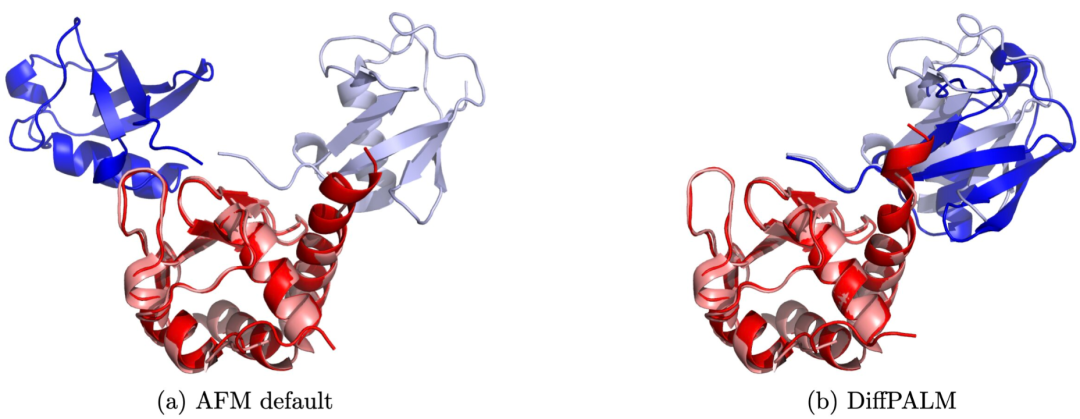
Kurseinführung:1. Bedeutung von Proteininteraktionen Proteine sind die Bausteine des Lebens und an nahezu allen biologischen Prozessen beteiligt. Um die Komplexität der Zellfunktion zu erklären, ist es wichtig zu verstehen, wie Proteine interagieren. 2. Neue Methode: Paarung interagierender Proteinsequenzen Anne-Florence Bitbols Team an der Ecole Polytechnique Fédérale de Lausanne (EPFL) schlug eine Methode zur Paarung interagierender Proteinsequenzen vor. Diese Methode nutzt die Leistungsfähigkeit von Proteinsprachmodellen, die auf mehreren Sequenzausrichtungen trainiert wurden. 3. Vorteile der Methode Diese Methode eignet sich gut für kleine Datensätze und kann die Strukturvorhersage von Proteinkomplexen durch überwachte Methoden verbessern. 4. Die Forschungsergebnisse werden unter dem Titel „Pairinginteractingprotein“ veröffentlicht
2024-07-16 Kommentar 0 843
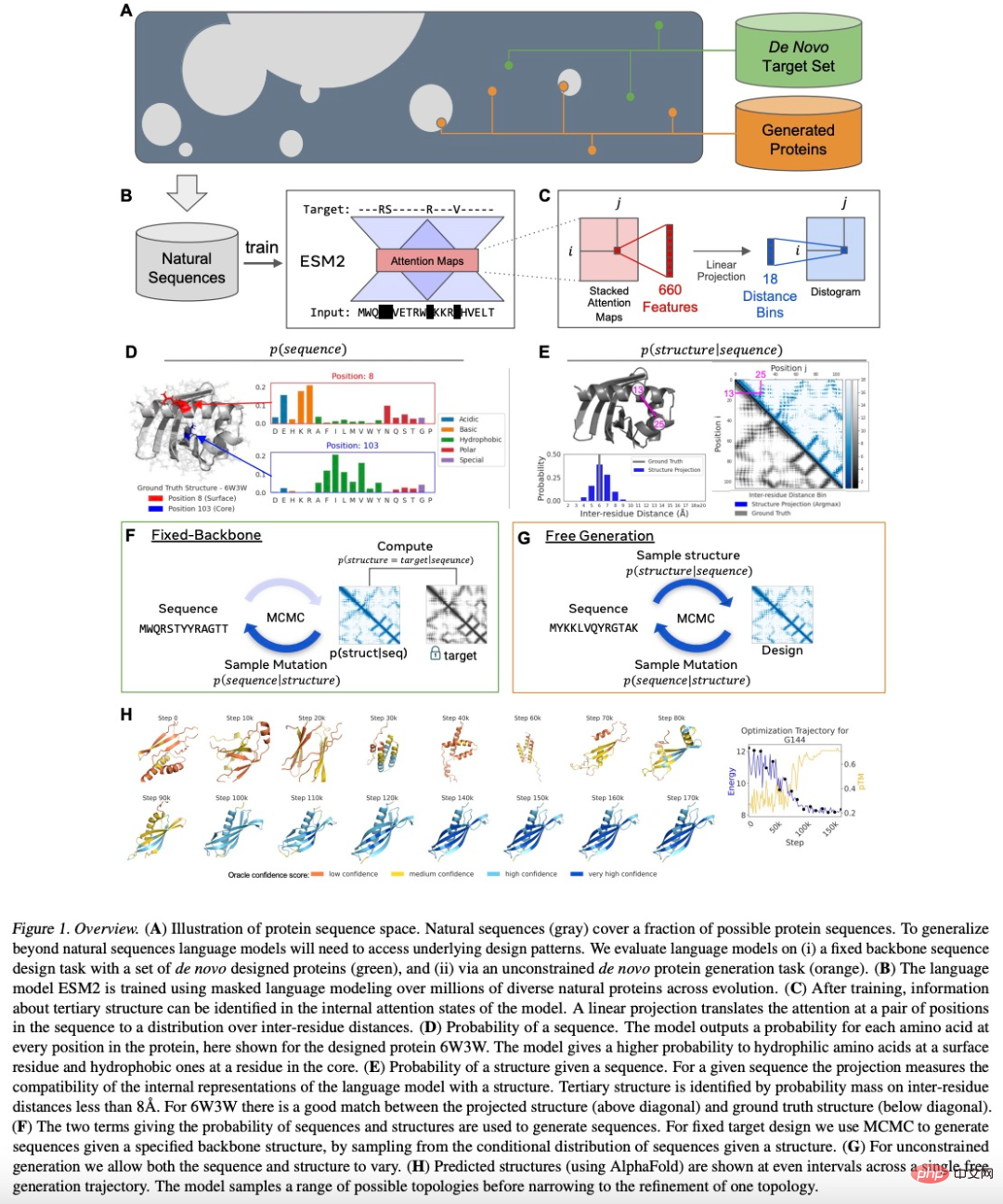
Kurseinführung:目录Sprachmodelle verallgemeinern über natürliche Proteine hinausEine High-Level-Programmiersprache für generatives ProteindesignDOC: Verbesserung der Kohärenz langer Geschichten mit detaillierter Gliederungskontrolle Skalierbare Diffusionsmodelle mit Transformern Point-E: Ein System für Gen
2023-04-13 Kommentar 0 1767

Kurseinführung:Herausgeber |. Das Design und die Entwicklung von Radish Skin Protein schreiten dank der Fortschritte im Deep Learning in beispiellosem Tempo voran. Aktuelle Modelle können jedoch nicht auf natürliche Weise Nicht-Protein-Entitäten während des Designprozesses berücksichtigen. Hier schlagen Forscher der Ecole Polytechnique Fédérale de Lausanne (EPFL) in der Schweiz eine Deep-Learning-Methode vor, die vollständig auf einem geometrischen Transformator von Atomkoordinaten und Elementnamen basiert und Proteine basierend auf Grundgerüsten mit Einschränkungen durch unterschiedliche molekulare Umgebungen vorhersagen kann. Mit dieser Methode können Forscher hoch thermostabile, katalytisch aktive Enzyme mit hoher Erfolgsquote herstellen. Es wird erwartet, dass dies die Vielseitigkeit von Proteindesign-Pipelines zur Erreichung gewünschter Funktionen erhöht. Diese Studie verwendet „Context-awaregeometricde“.
2024-08-05 Kommentar 0 941