

Herausgeber |. ScienceAI
Kürzlich schlug das Forschungsteam unter der Leitung von Zhang Haicang vom Institut für Computertechnologie der Chinesischen Akademie der Wissenschaften CarbonNovo vor, das Protein gemeinsam zu entwerfen Backbone-Struktur und -Sequenz durchgängig.
Diese Forschung wurde auf der Machine-Learning-Konferenz ICML 2024 unter dem Titel „CarbonNovo: Joint Design of Protein Structure and Sequence Using a Unified Energy-based Model“ veröffentlicht.

Hintergrundeinführung
Proteine sind wichtige Makromoleküle für biologische Funktionen. Das De-novo-Proteindesign zielt auf die Schaffung völlig neuer Proteine ab und findet breite Anwendung in der Arzneimittelentwicklung und Enzymtechnik.
In den letzten Jahren hat sich das KI-basierte Protein-De-novo-Design rasant entwickelt und wurde erfolgreich in Bereichen wie dem Antikörperdesign und dem Design kleiner Proteinmedikamente eingesetzt. Im Vergleich zu herkömmlichen Designmethoden hat es die Erfolgsquote und Effizienz des Designs erheblich verbessert.
KI-Proteindesign hat in den letzten Jahren von zwei großen technologischen Durchbrüchen profitiert:
Das erste ist das AlphaFold2-Modell im Bereich der Proteinstrukturvorhersage, das eine grundlegende neuronale Netzwerkmodellarchitektur und Proteinsequenz für den Bereich der Proteinberechnung bereitstellt. einschließlich Proteindesign- und Strukturdarstellungsmethoden sowie fortgeschrittener Trainingsstrategien (Destillationstraining, End-to-End-Training)
Zweitens die rasante Entwicklung von AIGC im Bereich Text, Bild und Video generation bietet ein ausgereiftes Generationsmodell für das Proteindesign, zum Beispiel DDPM, SDE, Flow Matching, Bayesian Flow Network usw. Die Hauptideen repräsentativer Proteindesignmodelle wie RFDiffusion und Chroma bestehen darin, diese beiden Technologien zu integrieren und das Proteinsequenz- und Strukturdarstellungsnetzwerk in ein KI-basiertes generatives Modellgerüst einzubetten.
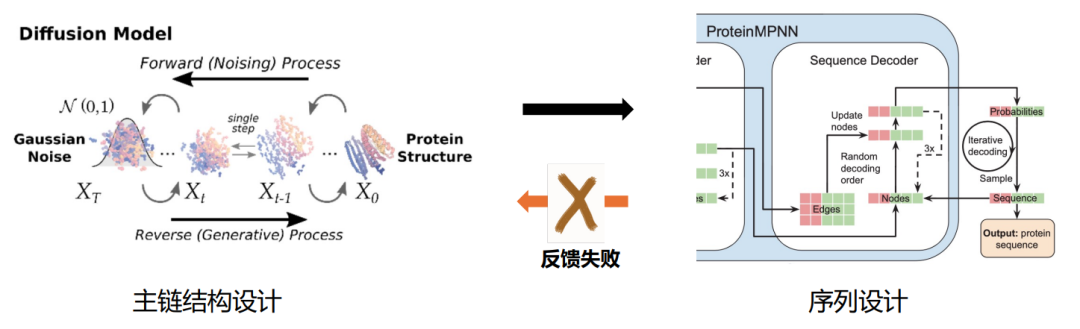
De-novo-Proteindesign umfasst hauptsächlich zwei Schritte: Design der Proteinrückgratstruktur und Sequenzdesign (Abbildung 1). Dementsprechend verwenden aktuelle Mainstream-Modelle normalerweise ein „zweistufiges“ Framework: Während des Trainingsprozesses werden das Strukturdesignmodul und das Sequenzdesignmodul separat trainiert, während im Inferenzprozess zuerst die Hauptkettenstruktur und dann die optimale Struktur generiert wird Die Hauptkettenstruktur wird generiert. Repräsentativ werden die beiden Softwareprogramme RFDiffusion und ProteinMPNN in der Praxis verwendet, um nacheinander die Struktur und Sequenz der Hauptkette zu generieren.
Das „zweistufige“ Framework für das De-novo-Proteindesign weist inhärente Einschränkungen auf:
CarbonNovo führt ein durchgängiges gemeinsames Design von Struktur und Sequenz durch
Als Reaktion auf die Einschränkungen des „zweistufigen“ Rahmenwerks für das Proteindesign hat ein Forschungsteam unter der Leitung von Zhang Haicang vom Institute of Computing Technology, Die Chinesische Akademie der Wissenschaften schlug CarbonNovo vor, gemeinsam die Strukturen der Proteinhauptkette durchgängig und sequenziell zu entwerfen. Das Papier wurde kürzlich auf der Machine-Learning-Konferenz ICML 2024 veröffentlicht.
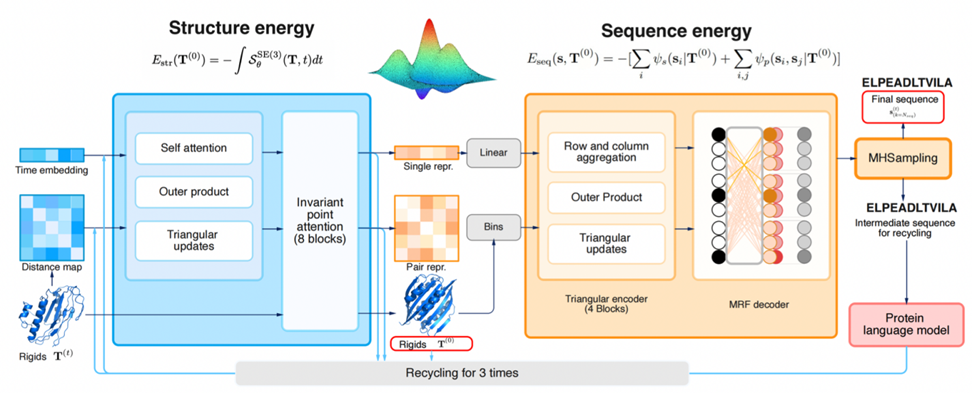
Abbildung 2: CarbonNovo generiert End-to-End-Proteinstrukturen und -sequenzen. (Quelle: Papier)
Die wichtigsten Beiträge von CarbonNovo lassen sich wie folgt zusammenfassen:
Gemeinsames Energiemodell der Proteinstruktur-Sequenz
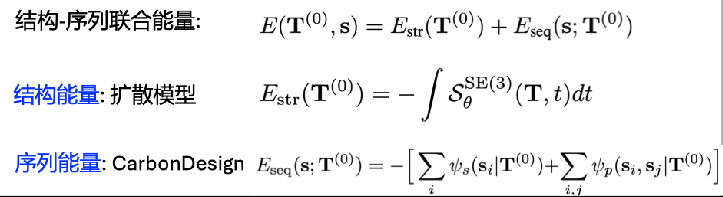
在经典物理模型下,天然蛋白质构象具有比较低的自由能,这也是蛋白质结构预测和设计的一般假设。基于此,CarbonNovo 建立了蛋白质结构和序列的联合能量模型:
图 2 展示了 CarbonNovo 的具体生成过程:
CarbonNovo 生成蛋白质结构-序列的性能评测

论文采用多种指标充分评价了 CarbonNovo 在蛋白质从头设计方面的性能 (图 3),例如,可折叠性、多样性、新颖性是领域内常用的评价指标。此外,论文还采用了 Rosetta 能量和语言模型下的似然概率(Sequence plausibility)作为评价指标。
CarbonNovo 和当前主流的「两阶段」设计模型做了比较,例如 RFdiffusion, Chroma, Genie, FrameDiff 和 FrameFlow。在最关键的可折叠性指标上 CarbonNovo 显著超过了所有基线方法,在其他指标上也显著超过基线方法或者和基线方法相当。
为了展示 CarbonNovo 在联合设计序列与结构方面的优势,作者还对比了使用 ProteinMPNN 生成序列的结果(图 3 a-c)。可以观察到,联合设计模型可以设计出更加匹配的蛋白质主链结构和序列。

作者进一步评估了 CarbonNovo 在不同长度的蛋白质设计上的性能(图 4)。在设计比较短的蛋白质时(例如长度 100),各个模型的表现相当。而随着蛋白质长度增长,CarbonNovo 的设计性能显著优于「两阶段」设计模型。
消融实验

作者训练了多个消融模型,以评估关键组件对 CarbonNovo 性能的相对贡献(图 5)。语言模型、序列设计模块和辅助训练损失对 CarbonNovo 的性能都有贡献。其中,语言模型的引入表现出最显著的贡献。此外,使用基于能量的序列设计模块相比自回归模型,也能显著提升序列设计的性能。
Case study:蛋白质结构「插值」
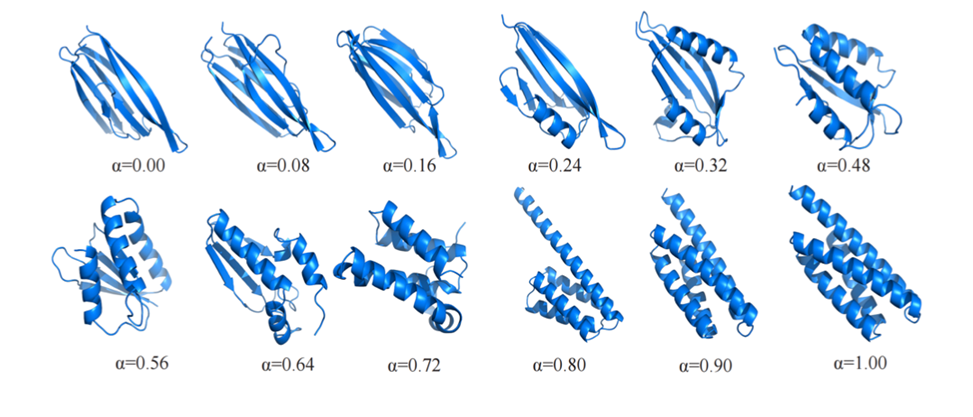
在图像生成领域,人脸图片插值/渐变是生成模型比较经典的应用。作者也尝试利用 CarbonNovo 进行蛋白质结构的插值。
图 5 展示了一个代表例子,随着在隐空间逐渐增加 all alpha-helices 结构向量的权重,生成的 all beta-sheets 结构会逐渐过渡到 all alpha-helices 结构。
这是领域内第一个关于蛋白质结构的插值实验,也体现了 CarbonNovo 学习到的蛋白质隐空间比较紧致。
结语
最后作者指出,虽然 CarbonNovo 主要侧重于蛋白质单体设计,但它也可以很容易被扩展到蛋白质复合物设计和条件设计,如多肽设计、抗体设计等。
作者团队目前在跟生物实验团队合作,通过湿实验的方式验证 CarbonNovo 设计的蛋白质。
作者所在 CarbonMatrix 团队,长期致力于 AI 蛋白质设计和 AI 药物设计,正在建立生物大分子结构设计和预测的统一生成模型。
其研究成果多次发表在 ICML、NeurIPS 等顶级机器学习会议和 Nature Machine Intelligence、Nature Communications 等顶级学术期刊上,目前也在和生物实验室合作,积极推动 AI 模型在药物设计领域的产业化落地。
Das obige ist der detaillierte Inhalt vonEin Team vom Institut für Computertechnologie der Chinesischen Akademie der Wissenschaften schlug CarbonNovo vor, ein KI-basiertes End-to-End-De-novo-Design von Proteinstrukturen und -sequenzen.. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
 Thunder 7 Crack-Patch
Thunder 7 Crack-Patch Wozu dient der Roaming-Ordner?
Wozu dient der Roaming-Ordner? So lösen Sie das Problem, dass CAD nicht in die Zwischenablage kopiert werden kann
So lösen Sie das Problem, dass CAD nicht in die Zwischenablage kopiert werden kann vscode
vscode Kernelpanic-Lösung
Kernelpanic-Lösung Das neueste Ranking der zehn besten Börsen im Währungskreis
Das neueste Ranking der zehn besten Börsen im Währungskreis Win11 „Mein Computer' zum Desktop-Tutorial hinzugefügt
Win11 „Mein Computer' zum Desktop-Tutorial hinzugefügt In Word gibt es eine zusätzliche leere Seite, die ich nicht löschen kann.
In Word gibt es eine zusätzliche leere Seite, die ich nicht löschen kann.



