
1. 引言
空間組學已擴展了分子類別分析的範圍,但許多技術受到空間分辨率的限制。現有計算方法主要針對轉錄組學數據,缺乏對新興空間組學技術的適應性。
2. soScope 框架
北京航空航天大學和清華大學的研究人員提出了 soScope,這是一個統一的生成框架,旨在提高空間組學數據的質量和分辨率。
3. 技術原理
soScope 匯總來自組學、空間關係和圖像的多模態組織資訊。透過分佈先驗與組學特定建模的聯合推斷,輸出具有增強分辨率的組學譜。
4. 性能評估
soScope 在Visium、Xenium、spatial-CUT&Tag、slide-DNA/RNA-seq 等平台上的評估結果表明:
5. 擴展應用
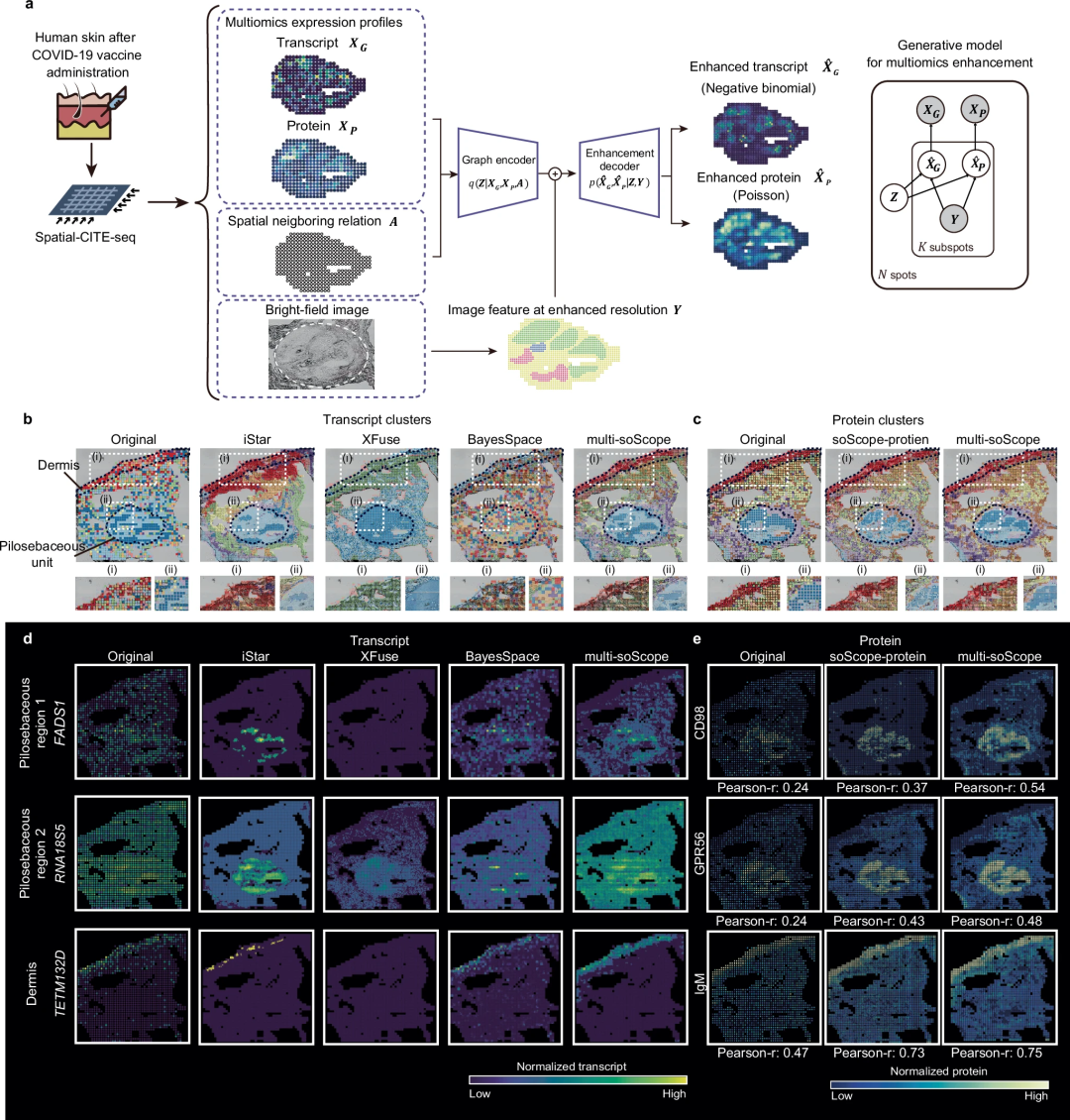
soScope 已擴展到spatial-CITE-seq 和空間ATAC-RNA-seq,利用跨組學參考組學增強。6. 結論
soScope 提供了一種多功能工具,提高了空間組學技術和資源的利用率。7. 參考
該研究已於 2024 年 8 月 2 日以「Tissue characterization at an enhanced resolution across spatial omics platforms with deep deep 捲髮為問題」發表。
挑戰和解決方案
儘管取得了早期成功,但空間組學技術仍面臨兩個主要挑戰:soScope:提高空間分辨率和數據品質
北航和清華的研究團隊引入了空間組學範圍(soScope),這是一個完全生成的框架,它模擬來自不同空間組學技術的點級概況的生成過程,旨在提高它們的空間解析度和資料品質。 soScope將每個點視為增強空間分辨率的「子點」的集合,其組學特徵與空間位置和形態模式相關。然後,soScope使用多模態深度學習框架整合斑點組學概況、空間關係和高分辨率形態學圖像,並聯合推斷子斑點分辨率下的組學概況。透過選擇組學特定分佈,soScope可以對不同的空間組學資料進行精確建模和減少變異。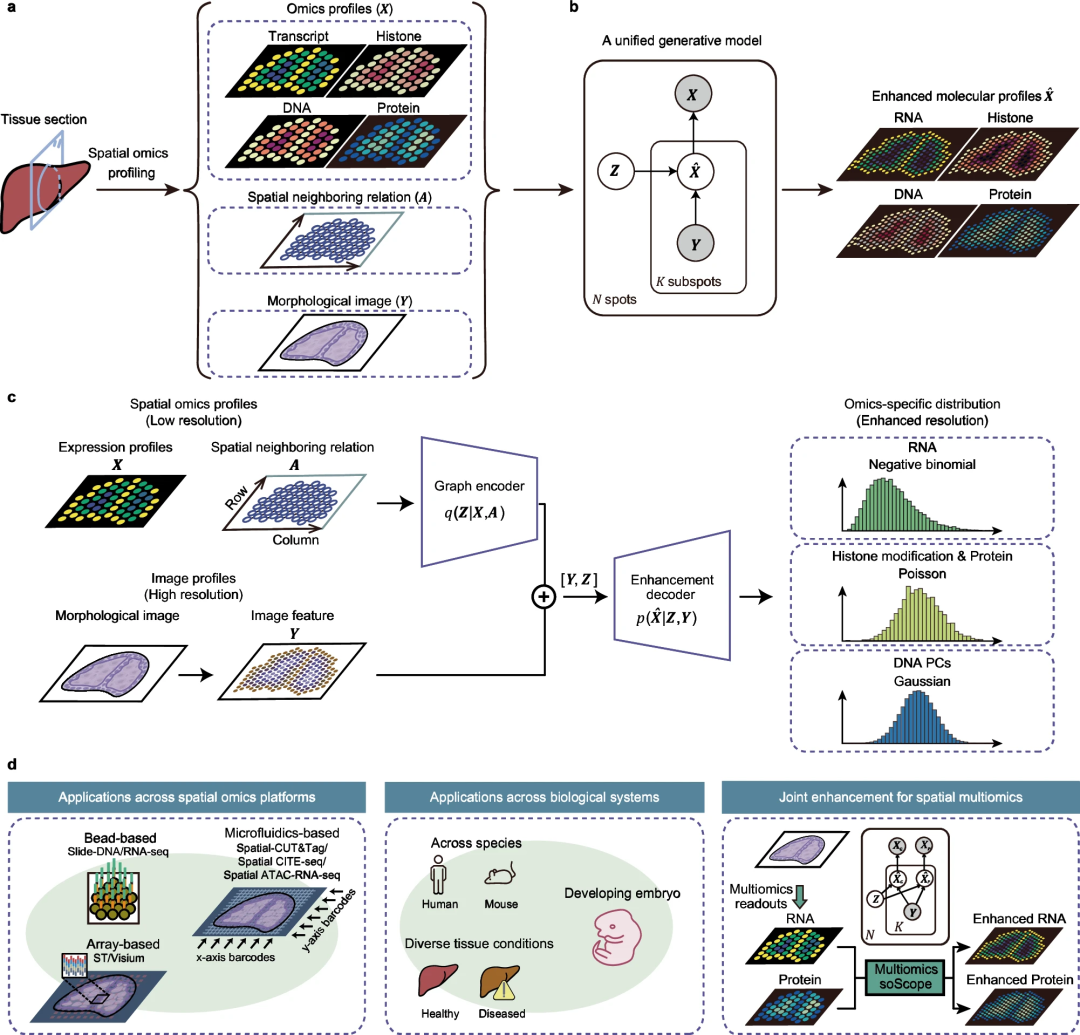
soScope 特點:
該團隊廣泛評估了soScope 對多種太空技術分析的多種分子類型的有效性和普遍性,包括Visium、Xenium、spatial-CUT&Tag、slide-DNA-seq、slide- RNA-seq、spatial-CITE-seq 和spatial ATAC-RNA-seq。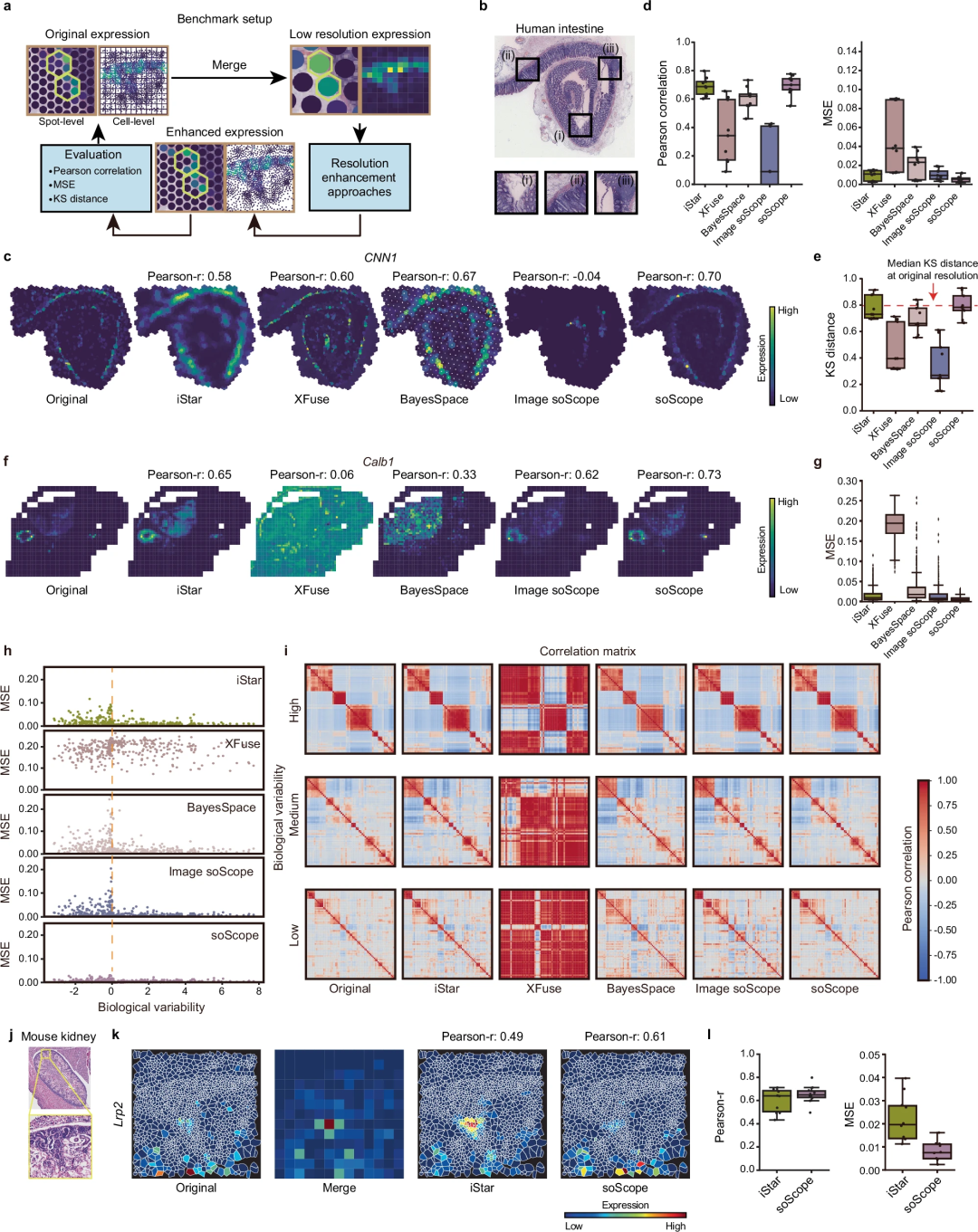
研究人員注意到,有幾種基於成像的空間組學技術,例如seqFISH、STARmap 和MERFISH,它們可以直接實現單細胞分辨率的空間分析,但代價是組學通量較低和組織區域較小。雖然 soScope 為預先指定的亞點或細胞位置提供了增強的配置文件,但它可能無法達到亞細胞分辨率。
進一步提高解析度:
降低計算成本:
對於包含來自同一器官的多個連續切片的較大數據集,soScope 可以:
潛力:
隨著空間組學資料資源的不斷擴展和新空間技術的出現,研究人員相信 soScope 具有以下潛力:
論文連結:
https://www.nature.com/articles/s41467-024-50837-5
以上是比原始解析度高36倍,北航、清華團隊用AI在多空間組學平台上高解析度表徵組織,登Nature子刊的詳細內容。更多資訊請關注PHP中文網其他相關文章!




