
蛋白質參與了細胞組成、肌肉收縮、消化食物、識別病毒等眾多生物學功能。
為了設計出更好的蛋白質(包括抗體),科學家經常在不同位置反覆變異氨基酸(按一定順序排列組成蛋白質的單位),直到使蛋白質獲得所需的功能。
但胺基酸序列的數量比世界上的沙粒還要多,因此找到最佳蛋白質,進而找到最佳潛在藥物,通常難度很高。當面臨這項挑戰時,科學家通常會花費數百萬美元,並在微型化、簡化版的生物系統中進行測試。
「這需要大量的猜測和驗證。」史丹佛大學(Stanford University)化學工程助理教授兼Arc 研究所創新研究員Brian L. Hie 說,「許多智慧演算法的目標是消除其中的猜測。」
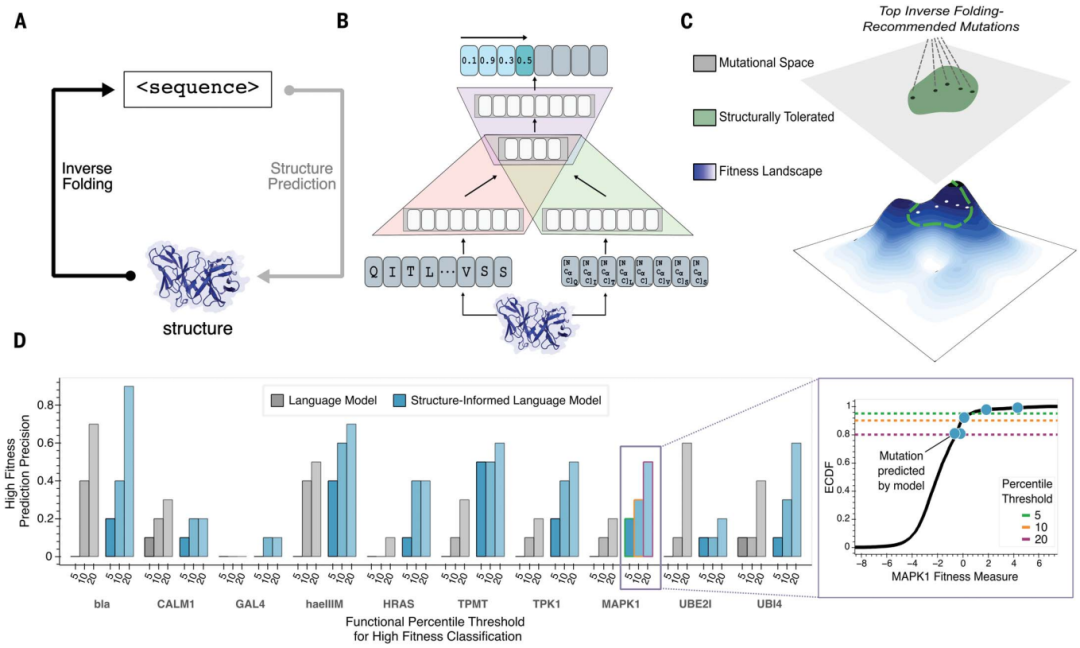
史丹佛大學的科學家開發了一種基於機器學習的新方法,可以更快、更準確地預測導致更好抗體藥物的分子變化。研究人員將蛋白質骨架的 3D 結構與基於胺基酸序列的大型語言模型結合,能夠在幾分鐘內找到罕見且理想的突變。
研究以「Unsupervised evolution of protein and antibody complexes with a structure-informed language model」為題,於 2024 年 7 月 4 日發佈在《Science》。

僅基於序列資訊進行訓練的大型語言模型可以學習蛋白質設計的高級原理。然而,除了序列之外,蛋白質的三維結構也決定了它們的特定功能、活性和可進化性。
針對抗體工程問題,史丹佛大學的研究人員應用結構資訊蛋白質語言模型,來預測受已知抗體或抗體-抗原複合物結構約束的高適應度序列。
研究表明,增強蛋白質結構主幹座標的通用蛋白質語言模型可以指導不同蛋白質的演化,而無需對單一功能任務進行建模。
結構引導範式:
廣泛應用:
蛋白質複合物設計:
人類抗體演化:
取代大量數據:
定向進化:

透過此方法,該團隊篩選了約30 種用於治療嚴重急性呼吸症候群冠狀病毒2 (SARS-CoV-2) 感染的兩種治療性臨床抗體的變體。同時,研究人員對 BQ.1.1 和 XBB.1.5 抗體逃脫病毒變異體的中和作用分別提高了 25 倍,親和力提高了 37 倍。
總之,這項工具將有助於快速應對新出現或正在發展的疾病。它還降低了製造更有效藥物的門檻。更強的藥物意味著需要更低的劑量,這意味著給定的劑量可以使更多的患者受益。
Paper link: https://www.science.org/doi/10.1126/science.adk8946
Related reports: https://phys.org/news/2024-07-ai-approach-optimizes- antibody-drugs.html
以上是登Science,藥物親和力增加37倍,AI對蛋白、抗體複合物進行無監督優化的詳細內容。更多資訊請關注PHP中文網其他相關文章!




