
Dalam beberapa tahun kebelakangan ini, dengan perkembangan pesat teknologi sel tunggal, kami telah dapat mengukur pelbagai ciri sel tunggal untuk mendapatkan data multi-modal sel tunggal (seperti scRNA-seq, scATAC-seq , Patch-seq) .
Data ini membantu kami memperoleh pemahaman yang mendalam tentang fungsi selular dan mekanisme molekul. Sebagai contoh, penyelidik baru-baru ini menggunakan kaedah pembelajaran mesin untuk menganalisis hubungan antara data multi-modal sel tunggal untuk memahami mekanisme biologi yang terlibat dalam jenis dan penyakit sel.
Walau bagaimanapun, pemerolehan data multi-modal sel tunggal selalunya mahal dan modaliti yang hilang sering berlaku. Kaedah pembelajaran mesin sedia ada biasanya memerlukan data berbilang modal yang dipadankan sepenuhnya untuk pengisian dan pembenaman data, dan tidak sesuai untuk situasi di mana modaliti tiada.
Untuk menyelesaikan masalah ini, makmal Wang Daifeng di Universiti Wisconsin-Madison telah membangunkan kaedah pembelajaran mesin sumber terbuka berdasarkan autoenkoder variasi bersama - Autoenkoder Variasi Bersama untuk Imputasi Multimodal dan Membenamkan (JAMIE).
JAMIE boleh digunakan untuk analisis penyepaduan data berbilang mod sel tunggal, seperti penjajaran data, pembenaman dan penambahan data yang hilang untuk meramalkan jenis dan fungsi sel dengan lebih baik.
Karya ini baru-baru ini diterbitkan dalam Nature Machine Intelligence.

Alamat kertas: https://www.nature.com/articles/s42256-023-00663 -z
Alamat projek: https://github.com/daifengwanglab/JAMIE
JAMIE melatih model autoenkoder variasi bersama yang boleh diguna semula untuk meningkatkan inferens corak mod tunggal dengan menayangkan data berbilang mod yang tersedia secara berasingan ke dalam keupayaan ruang terpendam yang serupa.
Seperti yang ditunjukkan dalam Rajah 1, untuk melakukan imputasi silang modal, JAMIE menyuap data ke dalam pengekod dan kemudian memproses hasil ruang terpendam melalui penyahkod bertentangan.
JAMIE menggabungkan penjanaan ruang terpendam yang boleh digunakan semula dan fleksibel bagi pengekod automatik dengan anggaran koresponden automatik kaedah penjajaran, membolehkan pemprosesan data berbilang mod dengan surat-menyurat yang tidak lengkap.
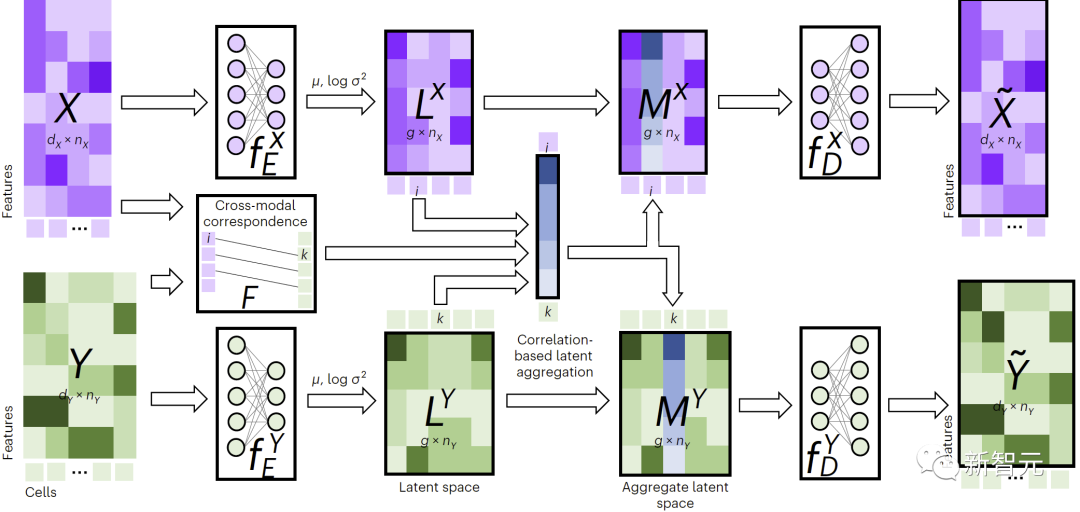
Rajah 1. Gambaran keseluruhan kaedah JAMIE
Secara khusus, JAMIE boleh dibahagikan kepada dua langkah berikut:
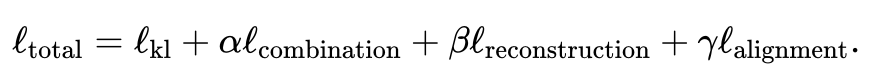
Jumlah fungsi kehilangan mengandungi empat item.
Item pertama mengira perbezaan Kullback-Leibler (KL) antara taburan yang disimpulkan oleh autoenkoder variasi dan taburan normal piawai multivariat, membantu mengekalkan ruang terpendam kesinambungan ruang.
Untuk ungkapan khusus setiap item, lihat teks asal kertas itu. Berat item kedua, ketiga dan keempat berbanding dengan item pertama boleh dilaraskan oleh pengguna juga menyediakan pemberat lalai yang sesuai untuk situasi biasa.
Jadual berikut menunjukkan perbandingan model dan skop JAMIE yang boleh digunakan dengan kaedah terkini. JAMIE menyatukan ciri beberapa kaedah integrasi dan interpolasi yang berbeza ke dalam seni bina tunggal, dengan itu membolehkan interpolasi modaliti yang hilang, membolehkan keserasian data bukan omik dan keupayaan untuk mengendalikan data berbilang mod dengan hanya separa kelebihan.
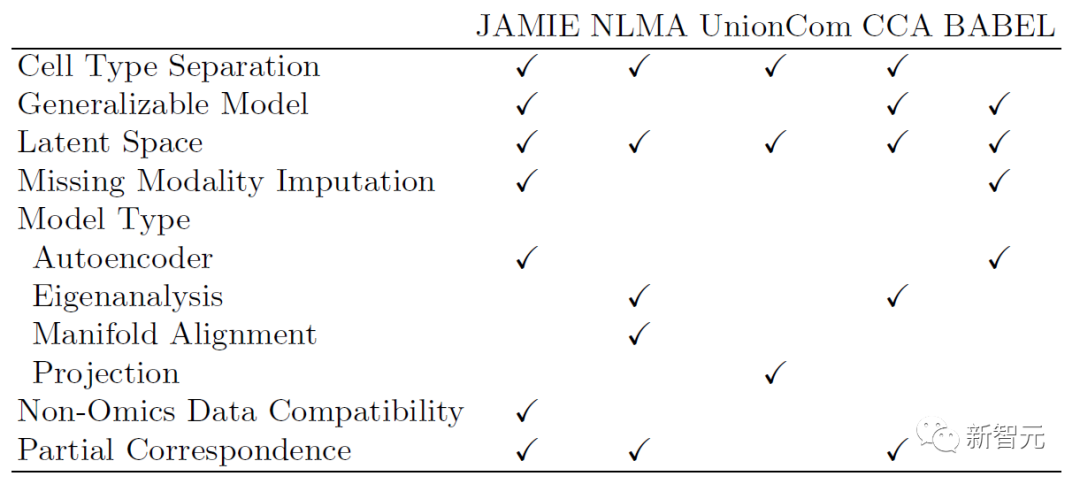
Jadual 1. Perbandingan pelbagai integrasi multimodal dan kaedah pengisian modal yang hilang. Melalui seni bina tunggal, JAMIE menyepadukan ciri daripada pelbagai kaedah penyepaduan dan interpolasi yang berbeza. NLMA: Penjajaran Manifold Tak Linear [15], UnionCom [7], CCA: Analisis Korelasi Kanonik [15, 16], BABEL [5].
Penyepaduan data berbilang mod dan ramalan fenotip
Pengintegrasian data multimodal boleh meningkatkan prestasi pengelasan, meningkatkan pengetahuan fenotip dan pemahaman tentang mekanisme biologi yang kompleks.
Memandangkan dua set data dan perhubungan yang sepadan, JAMIE boleh menjana data ruang terpendam, berdasarkan pengekod terlatih dan , dan melakukan pengelompokan atau pengelasan berdasarkan .
Pengelompokkan berdasarkan data ruang terpendam mempunyai beberapa kelebihan, seperti menggabungkan kedua-dua modaliti ke dalam penjanaan ciri. JAMIE kemudiannya boleh meramalkan surat-menyurat sampel, seperti ramalan jenis sel.
Untuk set data beranotasi separa, sel dalam kelompok yang sama harus mempunyai jenis yang serupa.
JAMIE memisahkan ciri jenis data yang berbeza dalam proses menjana data ruang terpendam, jadi algoritma pengelompokan atau pengelasan yang kompleks biasanya tidak diperlukan untuk mencapai hasil yang lebih baik.
Untuk data dimensi tinggi, JAMIE menggunakan UMAP [32] untuk visualisasi pengelompokan jenis sel.
Pengisian data rentas modal
Banyak kaedah pengisian data rentas modal semasa tidak boleh Tunjukkan bahawa mereka mempelajari mekanisme biologi asas untuk tujuan pelapik.
Berbanding dengan rangkaian suapan ke hadapan atau kaedah regresi linear, JAMIE boleh mempelajari mekanisme biologi asas dengan lebih baik untuk meramal data yang hilang berdasarkan asas matematik yang lebih ketat.
Rajah 2 menunjukkan proses JAMIE untuk pengisian data silang modal. JAMIE mula-mula melatih model pengekodan dan penyahkodan pada data latihan.
Untuk data baharu, JAMIE mula-mula menggunakan pengekod yang dipelajari daripada data untuk menayangkannya ke dalam ruang terpendam untuk mendapatkan , kemudian memperoleh dengan mengagregatkan ciri ruang terpendam, dan akhirnya menggunakan penyahkod yang sepadan untuk Data dinyahkodkan kepada corak yang hilang.
JAMIE menggunakan ruang terpendam untuk meramalkan korespondensi antara sel, yang mungkin membantu memahami hubungan antara ciri data dan fenotip.
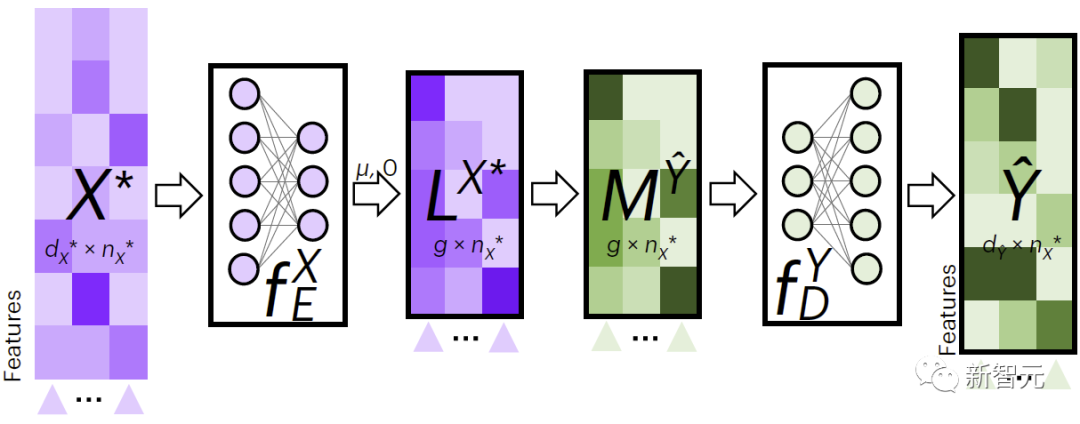
Rajah 2. JAMIE interpolasi silang modal
Penjelasan ciri ruang terpendam dan ciri padding
Untuk menerangkan model terlatih, JAMIE menggunakan SHAP (SHapley Additive expPlanations )[18].
SHAP menilai kepentingan ciri input individu dengan memodulasi sampel ramalan individu yang dijana oleh model. Ini boleh digunakan untuk pelbagai aplikasi yang menarik.
Jika pembolehubah sasaran boleh dipisahkan dengan mudah oleh fenotip, SHAP boleh mengenal pasti ciri-ciri yang berkaitan untuk kajian lanjut. Tambahan pula, jika kita melakukan imputasi, SHAP boleh mendedahkan sambungan silang modal yang dipelajari oleh model.
Memandangkan model dan sampel, pelajari nilai SHAP supaya di manakah vektor ciri latar belakang.
Jika , maka jumlah nilai SHAP dan output latar belakang akan sama dengan , di mana setiap satu adalah berkadar dengan kesan pada output model.
Satu lagi teknik berguna ialah memilih metrik utama untuk pengelasan (cth., LTA [7, 19]) atau imputasi (cth., surat-menyurat antara ciri yang dikira dan hubungan ciri yang diukur) dan menilai metrik dengan mengalih keluar (menggantikan dengan nilai latar belakang) setiap ciri satu demi satu dalam model.
Kemudian, jika metrik utama menjadi lebih teruk, ini menunjukkan bahawa ciri yang dialih keluar adalah lebih penting kepada keputusan model.
JAMIE menggunakan empat set data berbilang mod sel tunggal yang biasa digunakan untuk pengesahan.
(1) Data berbilang mod simulasi (300 sampel, 3 jenis sel) yang dijana oleh pensampelan taburan Gaussian bagi manifold bercabang daripada MMD-MA; >(2) Gen patch-seq daripada sel neuron tunggal dalam korteks visual tetikus (3,654 sampel, 6 jenis sel) dan korteks motor tetikus (1,208 sampel, 9 jenis sel) Data ekspresi dan pencirian elektrofisiologi; > (3) 8,981 sampel daripada otak manusia yang sedang berkembang (21 minggu kehamilan, 7 jenis sel utama yang meliputi korteks serebrum manusia) 10x ekspresi gen multi-omik sel tunggal dan data kebolehcapaian kromatin
(4) gen scRNA-seq daripada 4,301 sel daripada garisan sel adenokarsinoma kolon COLO-320DM Ekspresi dan data kebolehcapaian kromatin scATAC-seq.
Penilaian mendapati JAMIE jauh lebih baik daripada kaedah lain (perbandingan keputusan data simulasi manifold cawangan MMD-MA dalam Rajah 3, dan perbandingan hasil data korteks visual tetikus dalam Rajah 4 ) dan Ciri-ciri penting pengisian berbilang mod diutamakan sambil menyediakan cerapan mekanistik baharu yang berpotensi pada tahap resolusi selular.
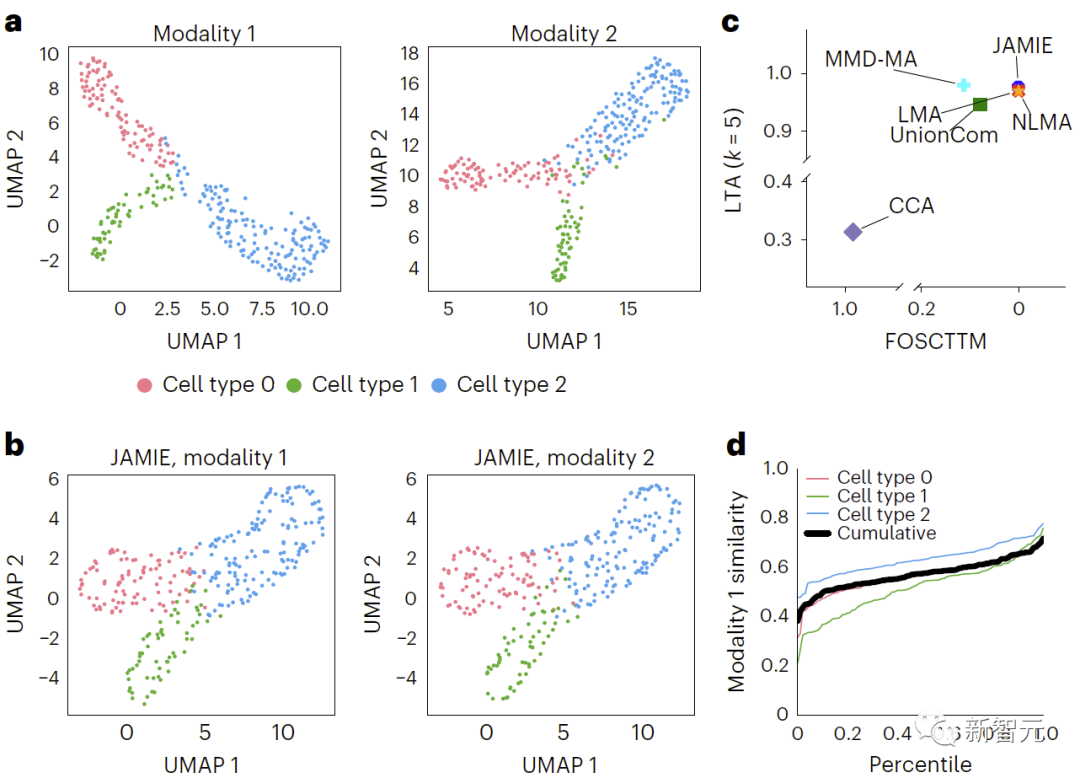
Dalam Rajah 3, dengan menggunakan algoritma UMAP pada data spatial mentah dan mewarna mengikut sel yang berbeza jenis , berbanding hasil simulasi data multimodal. b. UMAP ruang terpendam JAMIE. c. JAMIE dan teknik sedia ada (CCA[15,16], LMA[15], MMD-MA[8], NLMA[15] dan UnionCom[7]) apabila menggunakan semua maklumat surat-menyurat yang tersedia untuk pemisahan jenis sel. Paksi-x ialah perkadaran sampel yang lebih hampir kepada min sebenar, dan paksi-y ialah nilai LTA[7,19]. Dalam mod 1, taburan kumulatif jarak 1-JS dikira untuk menilai persamaan nilai yang diukur dan diinterpolasi. Setiap garis berwarna mewakili persamaan jenis sel tertentu, manakala garis hitam mewakili persamaan purata merentas jenis sel.
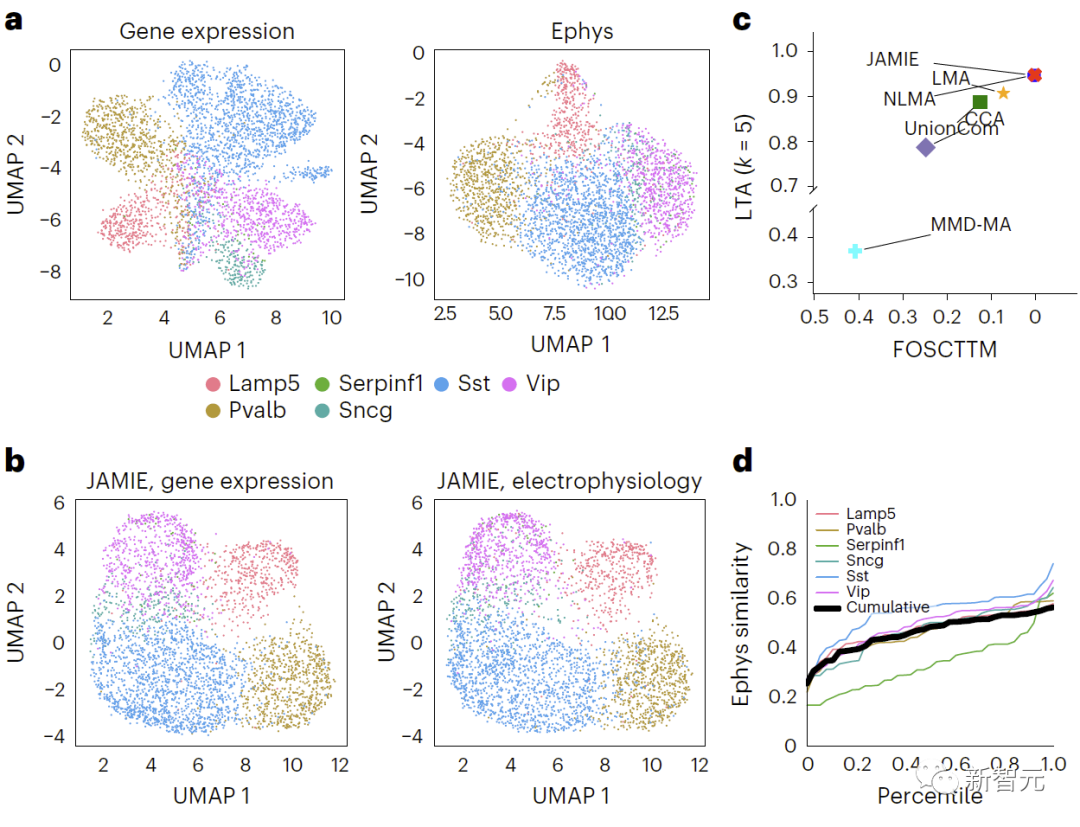
Dinyatakan semula: Membandingkan hasil ekspresi gen dan ciri elektrofisiologi dalam korteks visual tetikus, Berbeza jenis sel diwarnakan menggunakan UMAP dalam ruang asal. Rajah 4 menunjukkan keputusan perbandingan. b. UMAP ruang terpendam JAMIE. c. JAMIE dan teknik sedia ada (CCA[15,16], LMA[15], MMD-MA[8], NLMA[15] dan UnionCom[7]) apabila menggunakan semua maklumat surat-menyurat yang tersedia untuk pemisahan jenis sel. Paksi-x ialah perkadaran sampel yang lebih hampir kepada min sebenar, dan paksi-y ialah nilai LTA[7,19]. Dalam mod 1, taburan kumulatif persamaan antara nilai yang diukur dan diinterpolasi yang dikira dari jarak 1-JS dikaji. Setiap garis berwarna mewakili persamaan satu jenis sel, manakala garis hitam mewakili persamaan purata jenis sel yang berbeza.
Ringkasnya, JAMIE ialah model rangkaian neural dalam yang baru untuk ramalan bersepadu data berbilang mod sel tunggal.
Ia sesuai untuk data berbilang mod yang kompleks, bercampur atau separa sepadan, dilaksanakan melalui kaedah pengagregatan pembenaman terpendam baru yang bergantung pada struktur pengekod auto variasi bersama (VAE). Selain prestasi unggul yang dinyatakan di atas, JAMIE juga mempunyai keupayaan pengkomputeran yang cekap dan keperluan penggunaan memori yang rendah. Selain itu, model terlatih dan benam terpendam rentas mod yang dipelajari boleh digunakan semula dalam analisis hiliran.
Sudah tentu untuk set data yang lebih besar, latihan pengekod auto variasi (VAE) mengambil banyak masa. Oleh itu, kaedah pemilihan ciri sebelum ini seperti PCA automatik dalam JAMIE membantu mengurangkan keperluan masa. Memandangkan VAE menggunakan kehilangan pembinaan semula, prapemprosesan data juga penting untuk mengelakkan ciri besar atau berulang daripada menjejaskan ciri terbenam dimensi rendah secara tidak seimbang. Untuk imputasi rentas modal tertentu, kepelbagaian set data latihan mesti dipertimbangkan dengan teliti untuk mengelakkan berat sebelah model akhir dan memberi kesan negatif kepada keupayaan generalisasinya. JAMIE juga berpotensi diperluaskan untuk menyelaraskan set data daripada sumber yang berbeza dan bukannya modaliti yang berbeza, seperti data ekspresi gen dalam keadaan yang berbeza.
Pengarang kertas kerja ialah Noah Cohen Kalafut (pelajar PhD di Jabatan Sains Komputer), Huang Xiang (penyelidik kanan), dan Wang Daifeng (PI) bergabung dengan Jabatan Biostatistik dan Informatik Perubatan Universiti Wisconsin-Madison, Jabatan Sains Komputer dan Pusat Penyelidikan Weisman. Penulis yang sepadan ialah Profesor Wang Daifeng.
Ditubuhkan pada tahun 1973, Pusat Weisman telah memajukan penyelidikan dalam pembangunan manusia, gangguan perkembangan saraf dan penyakit neurodegeneratif selama setengah abad.
Atas ialah kandungan terperinci Kaedah analisis dan penjanaan data berbilang modal pasukan UW-Cina yang baharu JAMIE meningkatkan keupayaan ramalan jenis sel dan fungsi. Untuk maklumat lanjut, sila ikut artikel berkaitan lain di laman web China PHP!




