
단백질은 세포 구성, 근육 수축, 음식 소화, 바이러스 식별 등 많은 생물학적 기능에 관여합니다.
더 나은 단백질(항체 포함)을 설계하기 위해 과학자들은 종종 단백질이 필요한 기능을 얻을 때까지 여러 위치에서 아미노산을 반복적으로 돌연변이(단백질을 구성하는 단위를 특정 순서로 배열)합니다.
그러나 세상에 있는 모래알 수보다 더 많은 아미노산 서열이 있기 때문에 최고의 단백질, 즉 최고의 잠재적 약물을 찾는 것이 종종 어렵습니다. 이러한 문제에 직면했을 때 과학자들은 종종 수백만 달러를 지출하고 생물학적 시스템의 소형화되고 단순화된 버전을 테스트합니다.
“이에는 많은 추측과 검증이 필요합니다.” Stanford University의 화학 공학 조교수이자 Arc Institute의 혁신 펠로우인 Brian L. Hie는 다음과 같이 말했습니다. “많은 지능형 알고리즘의 목표는 추측을 없애는 것입니다.
스탠포드 대학교 과학자들이 더 나은 항체 약물을 더 빠르고 정확하게 만드는 분자 변화를 예측할 수 있는 기계 학습 기반의 새로운 방법을 개발했습니다. 단백질 백본의 3D 구조를 아미노산 서열을 기반으로 한 대규모 언어 모델과 결합하여 연구자들은 몇 분 안에 희귀하고 바람직한 돌연변이를 찾을 수 있었습니다.
이 연구의 제목은 "구조 정보 언어 모델을 사용한 단백질 및 항체 복합체의 비지도 진화"이며 2024년 7월 4일 "Science"에 게재되었습니다.

서열 정보로만 훈련된 대규모 언어 모델은 단백질 설계의 높은 수준의 원리를 학습할 수 있습니다. 그러나 서열 외에도 단백질의 3차원 구조가 단백질의 특정 기능, 활성 및 진화 가능성을 결정합니다.
항체 공학 문제의 경우, 스탠포드 대학의 연구원들은 구조적으로 정보를 갖춘 단백질 언어 모델을 적용하여 알려진 항체 또는 항체-항원 복합체 구조에 의해 제한되는 적합도가 높은 서열을 예측했습니다.
연구에 따르면 단백질 구조 백본 좌표로 강화된 범용 단백질 언어 모델은 개별 기능 작업을 모델링할 필요 없이 다양한 단백질의 진화를 안내할 수 있습니다.
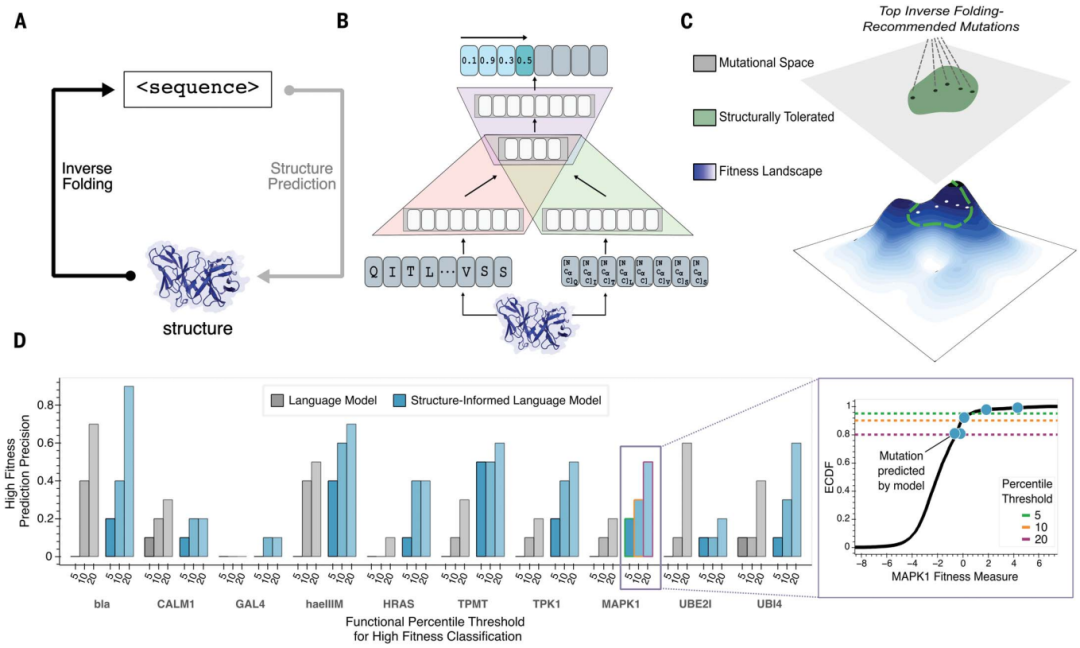
구조 기반 패러다임:
다양한 적용:
단백질 복합체 설계:
인간 항체 진화:
대량의 데이터 교체:
유도 진화:

이 방법으로 팀은 중증급성호흡기증후군 코로나바이러스 2(SARS-CoV-2) 감염 치료를 위한 2개의 치료 임상 항체 후보 약 30개를 선별했습니다. 동시에 연구원들은 BQ.1.1 및 XBB.1.5 항체 탈출 바이러스 변이체에 대한 중화율이 각각 25배 증가하고 친화력이 37배 증가했습니다.
결론적으로, 이 도구는 새로운 질병이나 진행 중인 질병에 신속하게 대응하는 데 도움이 될 것입니다. 또한 더 효과적인 약물을 만드는 데 대한 장벽을 낮춥니다. 더 강한 약물은 더 적은 용량이 필요하다는 것을 의미하며, 이는 더 많은 환자가 주어진 용량으로 혜택을 볼 수 있다는 것을 의미합니다.
논문 링크: https://www.science.org/doi/10.1126/science.adk8946
관련 보고서: https://phys.org/news/2024-07-ai-approach-optimizes- 항체약물.html
위 내용은 사이언스에 로그인, 약물 친화력 37배 증가, AI가 감독 없이 단백질 및 항체 복합체 최적화 수행의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!