
翻訳者|Cui Hao
レビュアー|Sun Shujuan
この記事では、科学者が医学における次の進歩を達成するのに TypeDB がどのように役立つかを検討し、有益なコード例を紹介します。結果を視覚的に示します。

#バイオテクノロジーの世界では、革新的な創薬に焦点を当てた誇大広告がたくさんあります。結局のところ、過去 10 年間はこの分野にとって黄金時代でした。過去 10 年間と比較して、2012 年から 2021 年の間に承認された新薬は 73% 増加し、これは前の 10 年間から 25% 増加しました。これらには、がんを治療する免疫療法、遺伝子療法、そしてもちろん、新型コロナウイルスワクチンが含まれます。こうした側面からも製薬業界が好調であることがわかります。
しかし、この傾向はますます憂慮すべきものになっています。創薬のコストとリスクは法外なものになってきています。これまで、新薬を市場に出すまでの平均コストは 10 億米ドルから 30 億米ドルで、平均期間は 12 年から 18 年です。同時に、新薬の平均価格は2007年の2000ドルから2021年には18万ドルまで高騰した。
だからこそ、多くの人が統計的機械学習などの人工知能 (AI) に期待を寄せ、早期の標的特定から治験まで新薬開発の加速に貢献しているのです。いくつかの化合物はさまざまな機械学習アルゴリズムを使用して同定されていますが、これらの化合物はまだ初期の発見または前臨床開発段階にあります。人工知能が創薬に革命をもたらすという期待は依然として刺激的ですが、実現されていません。
この約束を実現するには、人工知能が実際に何を意味するのかを理解することが重要です。近年、人工知能という用語は、あまり技術的な内容を含まない非常に一般的な用語になっています。では、本当の人工知能とは何でしょうか?
人工知能は学問分野として 1950 年代から存在し、時間の経過とともにさまざまな種類に分岐し、さまざまな学習スタイルを表しています。ペドロ・ドミンゴス教授は、著書『マスターズ・オブ・アルゴリズム: コネクショニスト、シンボリスト、進化論者、ベイジアン、シミュレーション主義者』の中で、これらのタイプ (彼はそれらを「部族」と呼んでいます) について説明しています。
過去 10 年間、ベイジアンとコネクショニストは世間の注目を集めてきましたが、シンボリストはそうではありませんでした。記号論は、論理的推論のための一連のルールに基づいて世界の現実的な表現を作成します。シンボリック AI システムは、他のタイプの AI ほど大きな知名度はありませんが、自動化された推論と知識表現という、他のタイプにはないユニークで重要な機能を備えています。
実際、知識表現の問題は創薬における最大の問題の 1 つです。リレーショナル データベースやグラフ データベースなどの既存のデータベース ソフトウェアは、生物学の複雑さを正確に表現して理解するのに苦労しています。
Drug Discovery によって定式化された問題は、さまざまな生物医学データ ソース (Uniprot や Disgenet など) の統合モデルを構築する必要性を示す好例です。データベース レベルでは、これは、タンパク質、遺伝子、薬物、病気、相互作用など、無数の複雑な実体と関係を記述するデータ モデル (これらをオントロジーと呼ぶ人もいるかもしれません) を作成することを意味します。
これが、オープンソース データベース ソフトウェアである TypeDB の目標です。開発者が、コンピュータが洞察を得るために使用できる非常に複雑なドメインの現実的な表現を作成できるようにすることです。
TypeDB の型システムはエンティティ関係の概念に基づいており、TypeDB に格納されているデータを表します。これにより、(型推論、ネストされた関係、ハイパーリレーション、ルール推論などを通じて) 複雑な生物医学領域の知識を取得するのに十分強力になり、科学者が洞察を得て医薬品開発時間を短縮できるようになります。
これは、セマンティック Web 標準を使用して疾病ネットワークをモデル化するのに 5 年以上苦労してきた大手製薬会社の例で示されていますが、TypeDB への移行後、わずか 3 週間で実装に成功し、この目標を達成しました。
たとえば、TypeQL (TypeDB のクエリ言語) で記述されたタンパク質、遺伝子、疾患を記述する生物医学モデルは次のようになります:
define protein sub entity, owns uniprot-id, plays protein-disease-association:protein, plays encode:encoded-protein; gene sub entity, owns entrez-id, plays gene-disease-association:gene, plays encode:encoding-gene; disease sub entity, owns disease-name, plays gene-disease-association:disease, plays protein-disease-association:disease; encode sub relation, relates encoded-protein, relates encoding-gene; protein-disease-association sub relation, relates protein, relates disease; gene-disease-association sub relation, relates gene, relates disease; uniprot-id sub attribute, value string; entrez-id sub attribute, value string; disease-name sub attribute, value string;
完全な動作例については、オープン ソースを見つけることができます。 Github 上の生物医学知識グラフ。これは、Uniprot、Disgenet、Reactome などのさまざまなよく知られた生物医学リソースからロードされています。
TypeDB に保存されたデータを使用して、次のような質問を行うクエリを実行できます。SARS ウイルスに関連する遺伝子と相互作用する薬剤はどれですか?
この質問に答えるには、TypeQL で次のクエリを使用します。
match $virus isa virus, has virus-name "SARS"; $gene isa gene; $drug isa drug; ($virus, $gene) isa gene-virus-association; ($gene, $drug) isa drug-gene-interaction;
これを実行すると、TypeDB はクエリ条件に一致するデータを返します。以下に示すように、TypeDB Studio で視覚化できます。これは、どの関連薬剤がさらなる調査に値するかを理解するのに役立ちます。
通过自动推理,TypeDB也可以推断出数据库中不存在的知识。这是通过编写规则来完成的,这些规则构成了TypeDB中模式的一部分。例如,一个规则可以推断出一个基因和一种疾病之间的关联,如果该基因编码的蛋白质与该疾病有关。这样的规则将被写成:
rule inference-example: when { (encoding-gene: $gene, encoded-protein: $protein) isa encode; (protein: $protein, disease: $disease) isa protein-disease-association; } then { (gene: $gene, disease: $disease) isa gene-disease-association; };
然后,如果我们要插入以下数据:
TypeDB将能够推断出基因和疾病之间的联系,即使没有插入到数据库中。在这种情况下,以下关系基因-疾病-关联将被推断出来。
match $gene isa gene, has gene-id "2"; $disease isa disease, has disease-name $dn; ; (gene: $gene, disease:$disease) isa gene-disease-assocation;
有了TypeDB对生物医学数据(符号)进行表示,再加上机器学习的上下文知识就可以让整个系统变得更加强大,从而增强洞察力。例如,可以通过药物探索管道发现有希望的目标。
寻找有希望的目标的方法是使用链接预测算法。TypeDB的规则引擎允许这样的ML模型执行,该模型通过推理推断对事实进行学习。这意味着从对平面的、无背景的数据学习转向对推理的、有背景的知识学习。其中一个好处是,根据领域的逻辑规则,预测可以被概括到训练数据的范围之外,并减少所需的训练数据量。
这样一个药物发现的工作流程如下:
1. 查询TypeDB,创建上下文知识的子图,利用TypeDB的全部表达能力。
2. 将子图转化为嵌入(embedding),并将这些嵌入到图学习算法中。
3. 预测结果(例如,作为基因-疾病关联之间的概率分数)可以被插入TypeDB,并用于验证/优先考虑某些目标。
有了数据库中的这些预测,我们可以提出更高层次的问题,利用这些预测与数据库中更广泛的背景知识。比如说:什么是最有可能成为黑色素瘤的基因目标,这些基因编码的蛋白质在黑色素细胞中如何表达?
用TypeQL写,这个问题看起来如下:
match $gene isa gene, has gene-id $gene-id; $protein isa protein; $cell isa cell, has cell-type "melanocytes"; $disease isa disease, has disease-name "melanoma"; ($gene, $protein) isa encode; ($protein, $cell) isa expression; ($gene, $disease) isa gene-disease-association, has prob $p; get $gene-id; sort desc $p;
这个查询的结果将是一个按概率分数排序的基因列表(如图学习者预测的):
{$gid "TOPGENE" isa gene-id;} {$gid "BESTGENE" isa gene-id;} {$gid "OTHERTARGET" isa gene-id;} ...
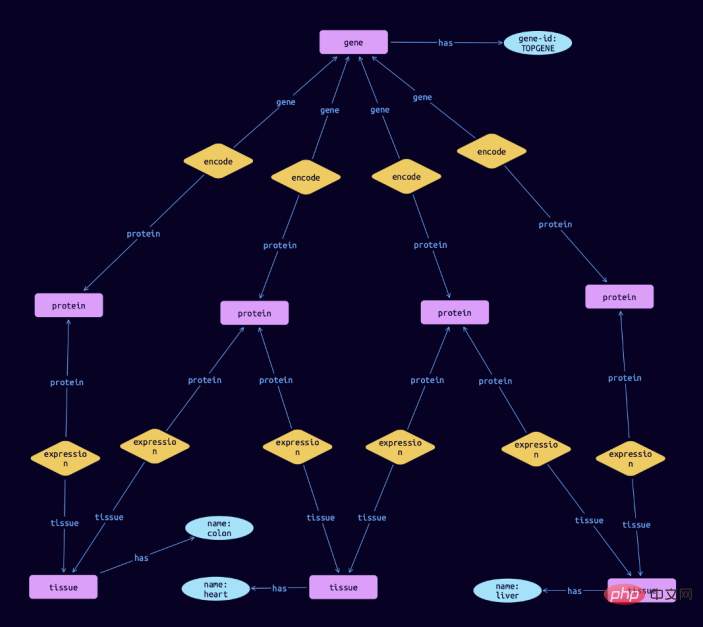
然后,我们可以进一步研究这些基因,例如通过了解每个基因的生物学背景。比方说,我们想知道TOPGENE基因编码的蛋白质所处的组织。我们可以写下面的查询。
match $gene isa gene, has gene-id $gene-id; $gene-id "TOPGENE"; $protein isa protein; $tissue isa tissue, has name $name; $rel1 ($gene, $protein); $rel2 ($protein, $tissue);
在TypeDB Studio中可视化的结果,可以显示这个基因编码的蛋白质在结肠、心脏和肝脏中的表达:
世界迫切需要创造治疗破坏性疾病的解决方案,希望通过人工智能的创新建立一个更健康的世界,在这个世界中每种疾病都可以被治疗。人工智能作用于药物探索仍处于起步阶段,但是如果一旦实现将会让生物学释放出新的创新浪潮,并使21世纪真正成为属于它的纪元。
在这篇文章中,我们看了TypeDB是如何实现生物医学知识的符号化表示,以及如何改善ML来为药物探索做出贡献的。在药物探索中应用人工智能的科学家们使用TypeDB来分析疾病网络,更好地理解生物医学研究的复杂性,并发现新的和突破性的治疗方式。
崔皓,51CTO社区编辑,资深架构师,拥有18年的软件开发和架构经验,10年分布式架构经验。
原文标题:Artificial Intelligence in Drug Discovery,作者:Tomás Sabat
以上が医療発見における人工知能の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。