
Editor | Violet
タンパク質、薬物、その他の生体分子間の相互作用は、さまざまな生物学的プロセスにおいて重要な役割を果たします。これらの相互作用を理解することは、生物学的プロセスの根底にある分子機構を解読し、新しい治療戦略を開発するために重要です。 タンパク質は細胞内で最も重要な分子の一つであり、細胞内でさまざまな機能を果たします。薬物は多くの場合、特定のタンパク質と相互作用することによって生理学的プロセスを調節します。これらの相互作用は、特定の分子シグナル伝達経路を促進または阻害する可能性があります。現在のマルチスケール計算方法では、単一のスケールに依存しすぎて、他のスケールが十分に適合していないことがよくあります。これは、不均一なマルチスケール指向性とマルチスケール学習に固有の貪欲さに関連している可能性があります。
最適化の不均衡を軽減するために、中山大学と上海交通大学の研究者は、学習のためにマルチスケール情報を効果的に統合できる、変数期待値最大化に基づくマルチスケール表現学習フレームワーク MUSE を提案しました。この戦略は、相互監視と反復最適化を通じて、原子構造と分子ネットワークスケール間のマルチスケール情報を効果的に融合します。このアプローチにより、より多くの情報伝達と学習が可能になります。この戦略は、相互監視と反復最適化を通じて、原子構造と分子ネットワークスケール間のマルチスケール情報を効果的に融合します。
MUSE+ は、分子相互作用 (タンパク質間、薬物間、薬物間) タスクに関して現在の最先端モデルよりも優れているだけでなく、タンパク質界面予測においても現在の最先端モデルよりも優れています。原子構造スケールで。さらに重要なことは、マルチスケール学習フレームワークは、他のスケールでの計算による創薬にも拡張できることです。
「
タンパク質と薬物の相互作用のバランスの取れたマルチスケール学習のための変分期待値最大化フレームワーク」と題されたこの研究は、5月25日付けの「Nature Communications」に掲載されました。
 論文リンク:
論文リンク:
これらの相互作用を純粋に構造から予測することは、構造生物学における最も重要な課題の 1 つです。現在の計算手法は主に分子ネットワークまたは構造情報に基づいて相互作用を予測しており、それらを統一されたマルチスケールのフレームワークに統合していません。
一部のマルチビュー学習方法はマルチスケール情報の融合を目指していますが、マルチスケール表現を学習する直感的な方法は、分子グラフと相互作用ネットワークを組み合わせて共同最適化することです。ただし、マルチスケール学習の不均衡と固有の貪欲さのため、これらのモデルは単一のスケールに大きく依存することがよくあります。あらゆる規模に関する情報を有効に活用できず、一般化が不十分である。
さらに、効果的なマルチスケール フレームワークは、さまざまなスケール内の豊富な情報を取得する必要があるだけでなく、それらの間の基礎的な関係を適切に保存する必要もあります。
タンパク質と薬物のマルチスケール情報を学習するための MUSEMUSE は、変分期待値最大化 (EM) フレームワークを通じて分子構造モデリングとタンパク質-薬物相互作用ネットワーク学習を組み合わせたマルチスケール学習方法です。 EM フレームワークは、複数の反復を交互に繰り返すプロセスで、2 つのモジュール、つまり期待ステップ (E ステップ) と最大化ステップ (M ステップ) を最適化します。
E ステップ中に、MUSE は各生体分子の構造情報を利用して、既知の相互作用と強化サンプルを使用した M ステップでのトレーニングに効果的な構造表現を学習します。これは、タンパク質と薬物のペアとそれらの原子レベルの構造情報を入力として受け取り、M ステップで予測される相互作用によって強化されます。 M ステップは、分子レベルの相互作用ネットワーク、構造埋め込み、および E ステップの予測相互作用を入力として受け取り、予測された相互作用を出力します。 E ステップと M ステップ間の反復最適化により、2 つのスケールで異なる学習率で分子構造とネットワーク情報を対話的に取得できます。
相互監視により、各スケール モデルが適切な方法で学習し、異なるスケールで効果的な情報を利用できるようになります。このフレームワークは、タンパク質と薬物間の相互作用の複数のマルチスケールにわたって実証されます。 MUSE はマルチスケール学習における特徴の不均衡を軽減し、異なるスケールからの階層的で補完的な情報を効果的に統合すると分析されています。
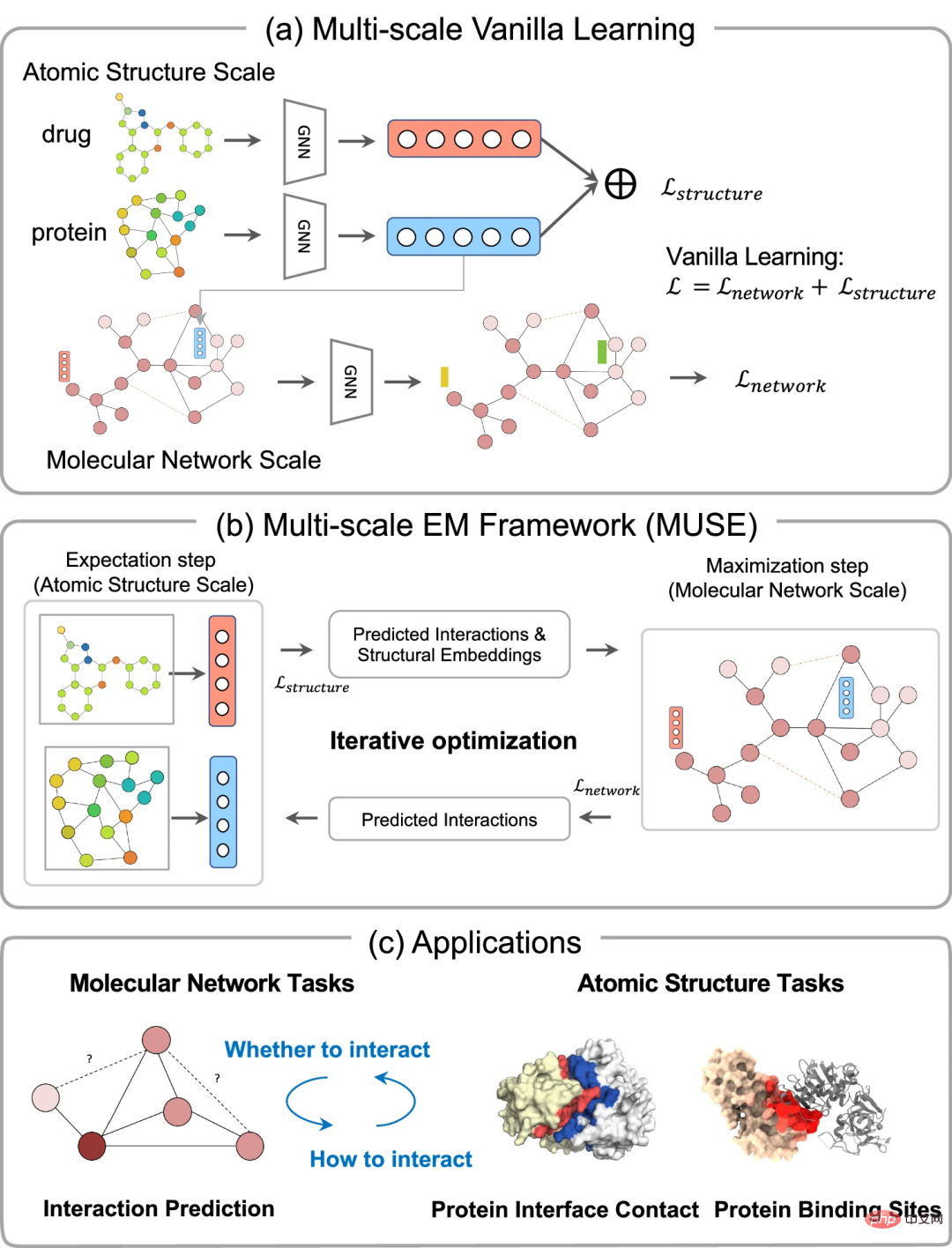
原子構造情報を利用して分子ネットワークスケールでの予測を改善します
彼らのアプローチを評価するには、まず、研究者らは、MUSE を使用して原子の構造情報を統合し、分子ネットワーク規模の予測を改善しました。 MUSE は、タンパク質間相互作用 (PPI)、薬物間相互作用 (DPI)、薬物間相互作用 (DDI) という 3 つのマルチスケール相互作用予測タスクで最先端のパフォーマンスを実現します。
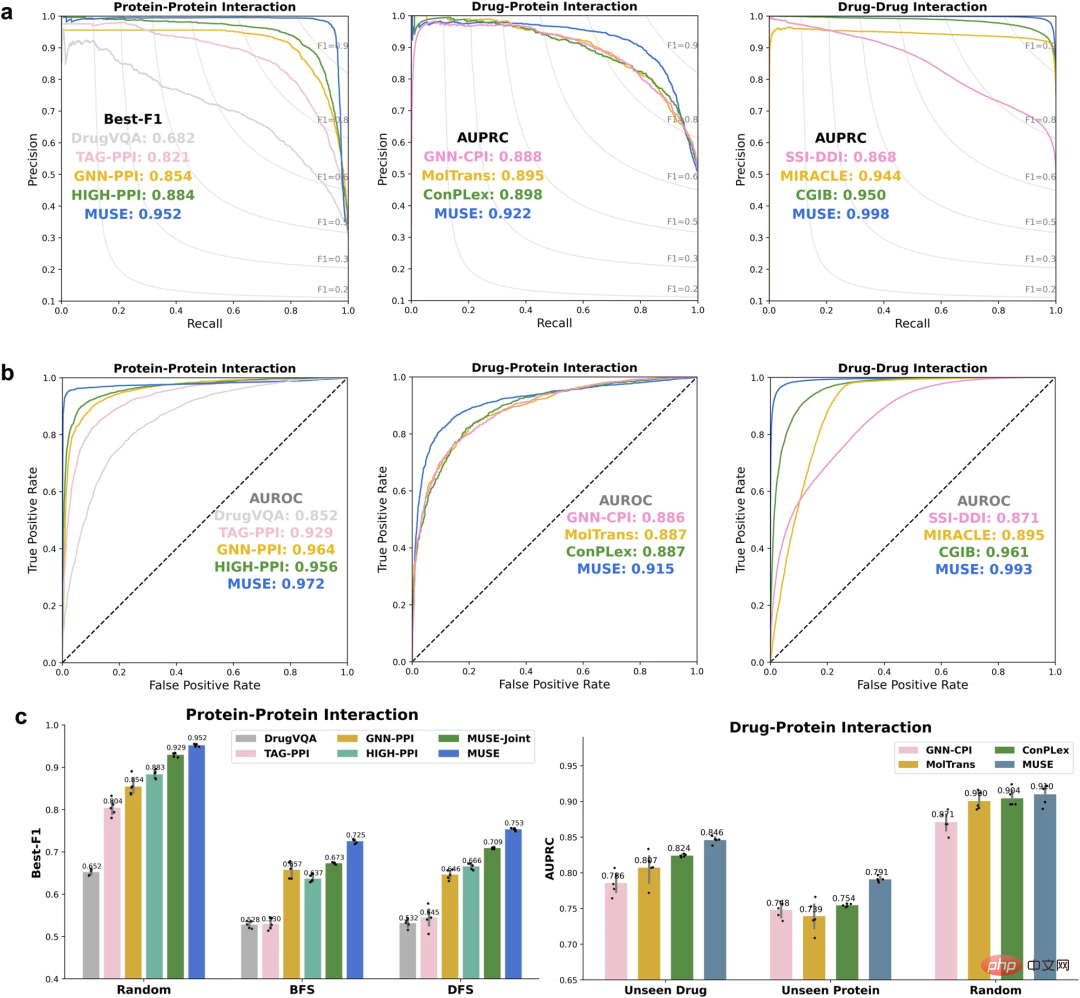
分子ネットワークスケールからの原子構造スケール予測の改善
研究者らは、原子構造情報を使用して分子ネットワークスケール予測を改善することに加えて、原子構造で構造を学習および予測するMUSEの能力をさらに研究しました。スケール PPI に関連する界面接触および結合部位の予測を含む、特徴付けを行う能力。
タンパク質の鎖間接触の予測を評価するために、MUSE を DIPS-Plus ベンチマークで最先端の手法と比較しました。 MUSE は他のすべての方法よりも一貫して優れており、原子構造予測におけるその有効性と適応性が検証されています。
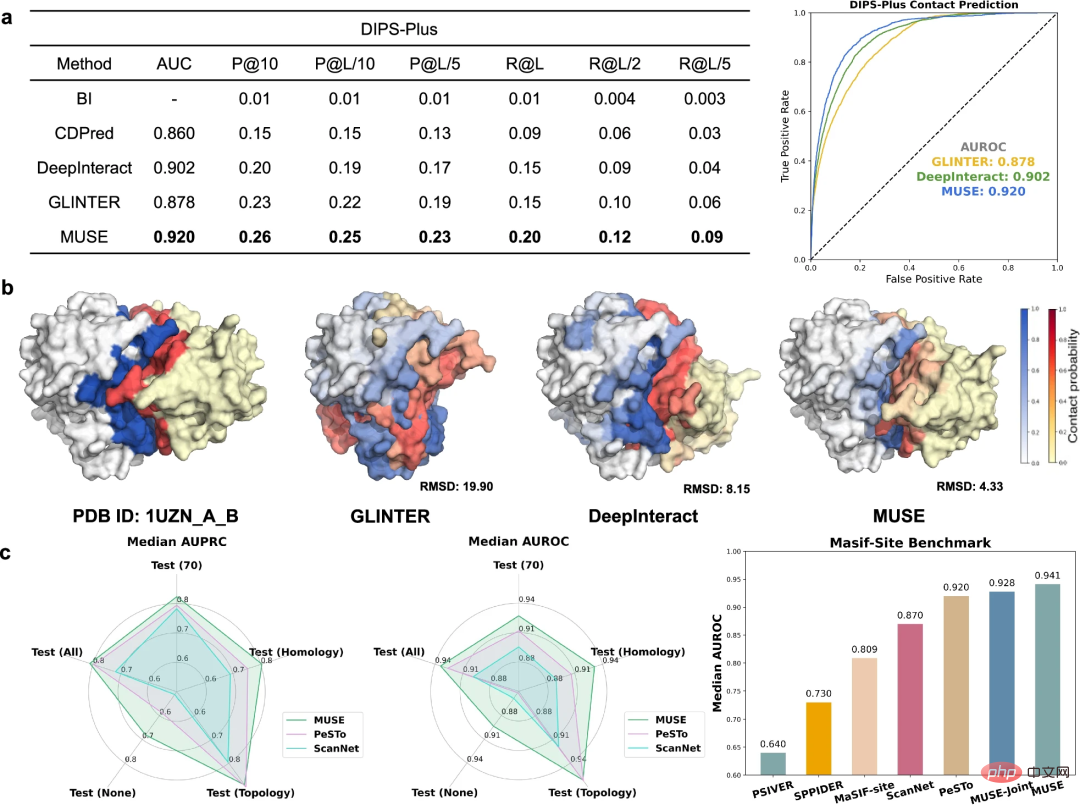
MUSE は、残基がタンパク質間相互作用に直接関与しているかどうかを予測するためにさらに評価されました。この結果は、MUSE での分子ネットワーク スケールの学習により、原子構造スケールの予測に対する貴重な洞察が得られることを示しています。
反復最適化によりマルチスケール学習の不均衡特性を緩和
なぜMUSEがマルチスケール表現において優れたパフォーマンスを達成できるのかを探るため、研究者らはマルチスケールの不均衡特性に基づいてMUSEの学習能力を分析しました。学ぶ。
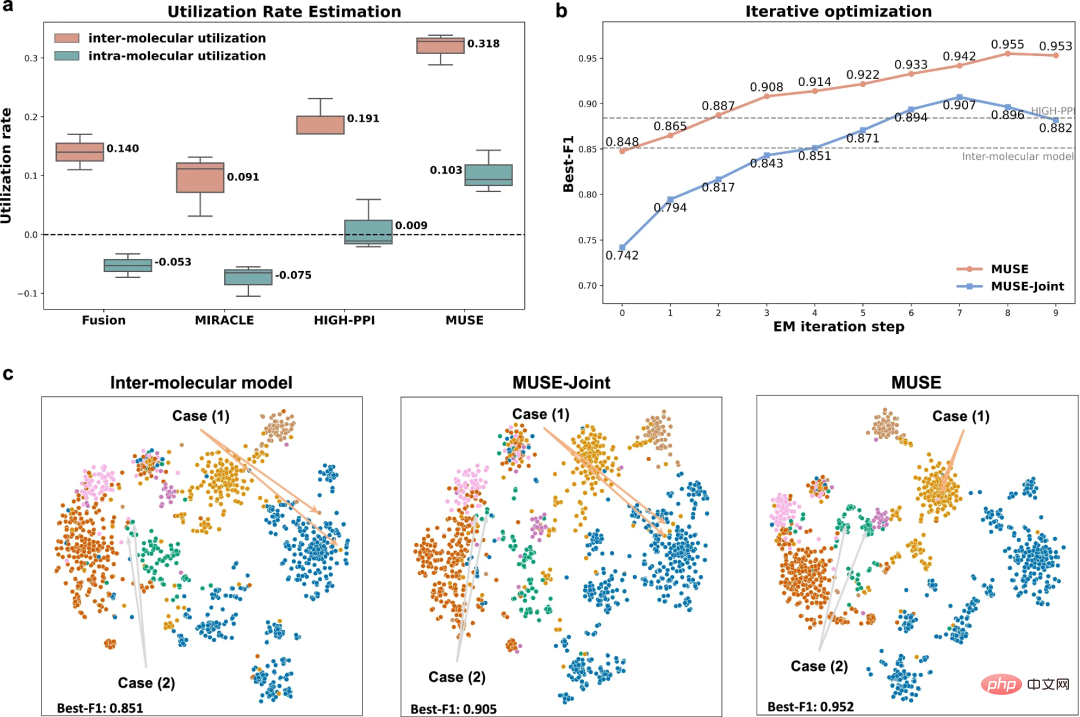
結果は、MUSE がマルチスケール学習における不均衡特性と貪欲な学習を効果的に軽減し、トレーニングプロセス中にさまざまなスケールでの情報の包括的な利用を保証することを示しています。さらに、使用率分析の実験により、研究者はモデルが学習した内容を具体的に理解できるようになり、MUSE を使用してさまざまなスケールでモデル学習のバランスをとれば汎化機能が強化できることが実証されました。
学習されたマルチスケール表現の視覚化と解釈
学習されたマルチスケール表現をよりよく理解するために、研究者は、MUSE によって学習されたマルチスケール表現をさまざまな角度から研究しました。(1) MUSE のキャプチャ アトミックの能力PPI に関与する構造情報 (つまり、構造モチーフと埋め込み)、(2) 学習された原子構造と分子ネットワーク表現の間の相互監視。
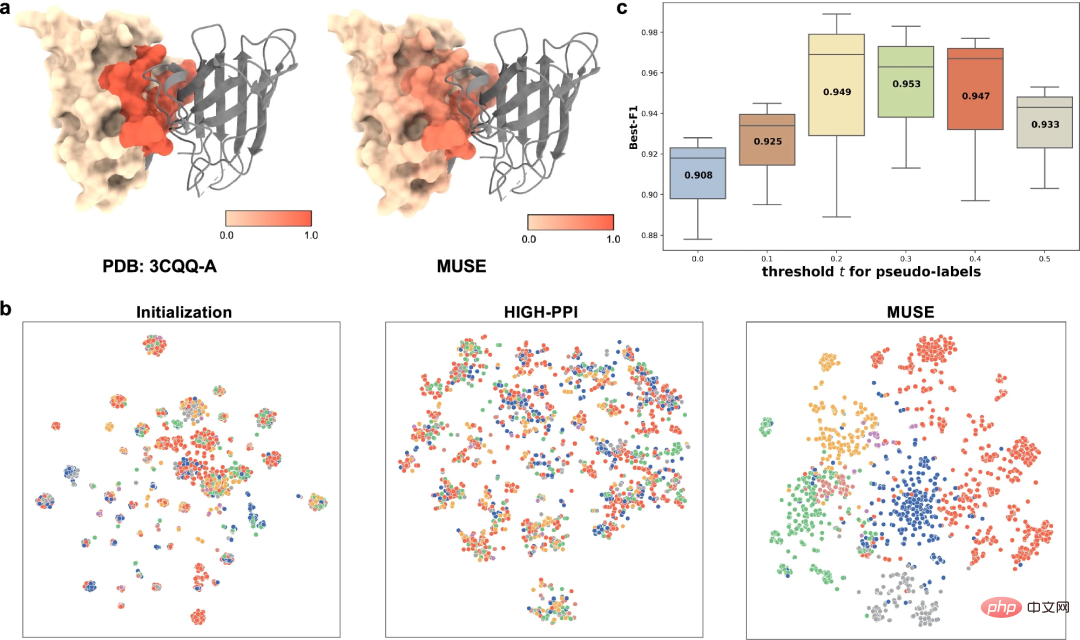
結合部位予測 (PDB id: 3CQQ-A) の例として、MUSE は結合部位に属する残基を 97.7% の精度で正確に識別できます。これは、MUSE における相互監視が、原子構造スケール モデルが相互作用に関連する重要な部分構造を学習するのに役立つことを示唆しています。
最後に、研究者らは、原子構造スケールで予測された擬似ラベルが分子ネットワークスケールに及ぼす影響を研究するために、アブレーション研究も実施しました。
MUSE はベンチマークで最先端のパフォーマンスを示していますが、ノイズが多く不完全なマルチスケールのダウンストリーム タスクを処理する能力を向上させることはまだ可能です。これは、ナレッジ グラフと説明可能な AI 技術による事前知識を組み合わせることで実現できます。一方、この概念的なマルチスケールのフレームワークは、他のスケールの計算による創薬にも拡張できます。
以上がSOTA パフォーマンス、マルチスケール学習、中山大学がタンパク質と薬物の相互作用 AI フレームワークを提案の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。