
Herausgeber |. Rettichhaut
Die Vorhersage der Protein-DNA-Bindungsspezifität ist eine anspruchsvolle, aber entscheidende Aufgabe, die zum Verständnis der Genregulation beiträgt. Protein-DNA-Komplexe binden typischerweise an ausgewählte DNA-Ziele, wohingegen Proteine an ein breites Spektrum von DNA-Sequenzen mit unterschiedlichem Grad an Bindungsspezifität binden. Diese Informationen sind nicht direkt in einer einzelnen Struktur zugänglich.
Um diese Informationen zu erhalten, haben Forscher der University of Southern California und der University of Washington den Deep Binding Specificity Predictor (DeepPBS) vorgeschlagen, ein geometrisches Deep-Learning-Modell zur Vorhersage der Bindungsspezifität basierend auf der Protein-DNA-Struktur.
DeepPBS kann interpretierbare Protein-Schweratom-Wichtigkeitswerte von Schnittstellenresten extrahieren. Diese Ergebnisse wurden durch Mutageneseexperimente validiert, wenn sie auf der Ebene der Proteinreste aggregiert wurden. Bei der Anwendung auf entworfene Proteine, die auf spezifische DNA-Sequenzen abzielen, konnte gezeigt werden, dass DeepPBS experimentell gemessene Bindungsspezifitäten vorhersagen kann.
Die Studie trug den Titel „Geometrisches Deep Learning der Protein-DNA-Bindungsspezifität“ und wurde am 5. August 2024 in „Nature Methods“ veröffentlicht.

Transkriptionsfaktoren regulieren Lebensprozesse durch Bindung an bestimmte DNA-Sequenzen. Dieser Bindungsmechanismus umfasst elektrostatische Wechselwirkungen, Desoxyribose-Stapelungseffekte und die Bildung von Wasserstoffbrückenbindungen.
Protein-DNA-Strukturinformationen werden normalerweise durch experimentelle Methoden wie Röntgenkristallographie, Kernspinresonanzspektroskopie oder Kryo-Elektronenmikroskopie gewonnen und in der Proteindatenbank (PDB) gespeichert. Diese Strukturen zeigen typischerweise gebundene DNA-Sequenzen und ihre physikalisch-chemischen Wechselwirkungen, decken jedoch nicht alle möglichen Bindungssequenzen ab.
Andererseits sind Hochdurchsatzexperimente wie Proteinbindungs-Microarrays, SELEX-seq usw. in der Lage, die Bandbreite potenzieller Bindungssequenzen zu erfassen, ihnen fehlen jedoch strukturelle Informationen.
Daher ist die Kombination von Strukturdaten und experimentellen Hochdurchsatzdaten von entscheidender Bedeutung, um die Bindungsspezifität von Transkriptionsfaktoren vollständig zu verstehen.
Derzeit bleibt die Vorhersage der Bindungsspezifität einer bestimmten Proteinsequenz innerhalb einer Proteinfamilie ein herausforderndes und ungelöstes Problem. Diese Schwierigkeit wird durch strukturelle Veränderungen im Bindungskontext und die große mechanistische Vielfalt verschärft.
„Die Struktur eines Protein-DNA-Komplexes enthält Proteine, die typischerweise an eine einzelne DNA-Sequenz binden. Um die Genregulation zu verstehen, ist es wichtig, die Bindungsspezifität eines Proteins an jede DNA-Sequenz oder Genomregion zu verstehen.“ Professor Remo Rohs von der University of Southern California.
In der neuesten Studie führten Forscher der University of Southern California und der University of Washington Binding Specificity Deep Predictors (DeepPBS) ein.
Rohs erklärte: „DeepPBS ist ein künstliches Intelligenztool, das Hochdurchsatzsequenzierung oder Strukturbiologieexperimente ersetzt, um die Protein-DNA-Bindungsspezifität aufzudecken.“
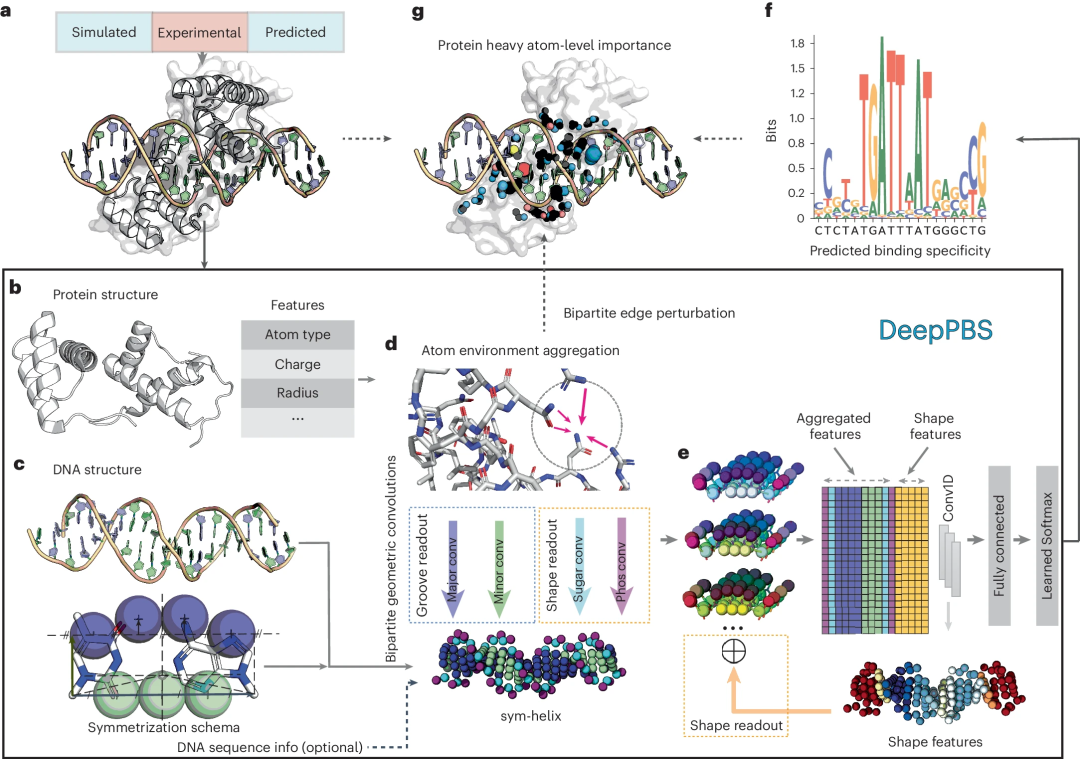
Dieses Deep-Learning-Modell zielt darauf ab, den physikalisch-chemischen und geometrischen Kontext von Protein-DNA-Wechselwirkungen zu erfassen, um die Bindungsspezifität vorherzusagen, ausgedrückt als Positionsgewichtsmatrix (PWM) basierend auf einer gegebenen Protein-DNA-Struktur. DeepPBS funktioniert über Proteinfamilien hinweg und dient als Brücke zwischen Strukturbestimmungs- und Bindungsspezifitätsbestimmungsexperimenten.
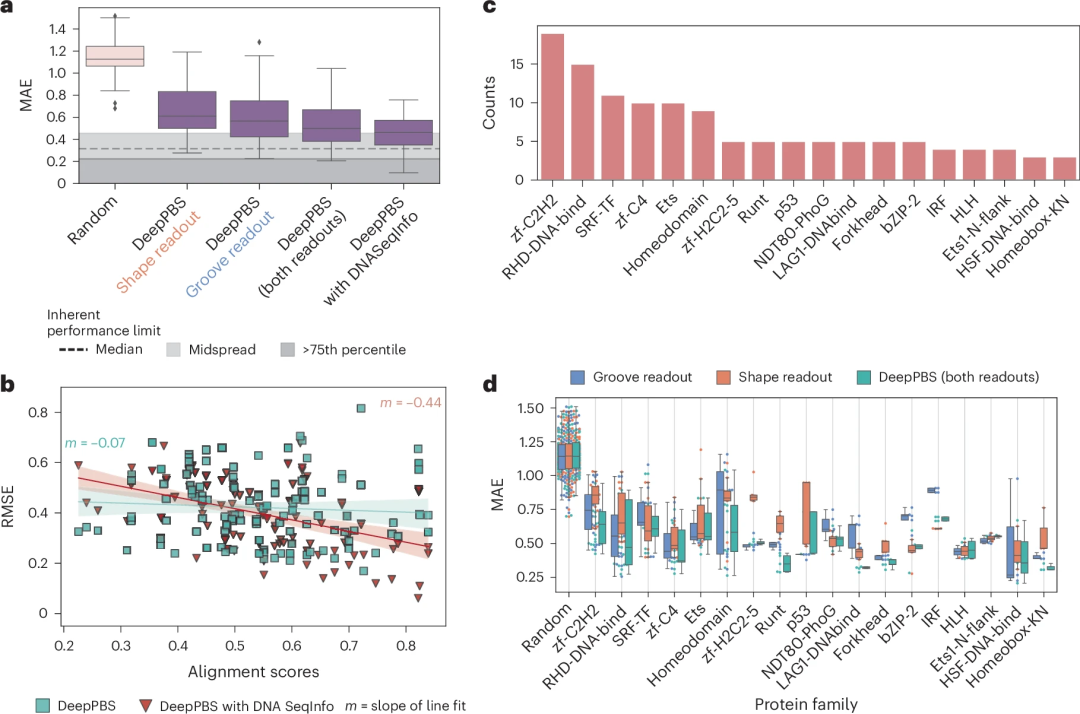
Abbildung: Leistung von DeepPBS zur Vorhersage der Bindungsspezifität über Proteinfamilien hinweg. (Quelle: Papier)
Eingaben in DeepPBS sind nicht auf experimentelle Strukturen beschränkt. Die rasante Entwicklung von Methoden zur Vorhersage der Proteinstruktur, darunter AlphaFold, OpenFold und RoseTTAFold, und Protein-DNA-Komplexmodellierern wie RoseTTAFoldNA (RFNA), RoseTTAFold All-Atom, MELD-DNA und AlphaFold3, hat dazu geführt, dass Strukturdaten für verfügbar sind Analyse Die Zahlen wachsen exponentiell.
Dieses Szenario verdeutlicht den wachsenden Bedarf an universellen Rechenmodellen zur Analyse von Protein-DNA-Strukturen. Die Forscher zeigen, wie DeepPBS in Verbindung mit Strukturvorhersagemethoden verwendet werden kann, um die Spezifität von Proteinen vorherzusagen, für die keine experimentelle Struktur verfügbar ist.
Darüber hinaus kann das Design von Protein-DNA-Komplexen verbessert werden, indem DeepPBS-Feedback genutzt wird, um die Bindung an DNA zu optimieren. Die Forscher zeigen, dass diese Pipeline eine vergleichbare Leistung wie ein aktuelles familienspezifisches Modell, rCLAMPS, aufweist, dabei aber allgemeiner ist: Insbesondere ist DeepPBS nicht auf Proteinfamilien beschränkt, kann mit biologischen Baugruppen umgehen und DNA-Seitenkettenpräferenzen vorhersagen.
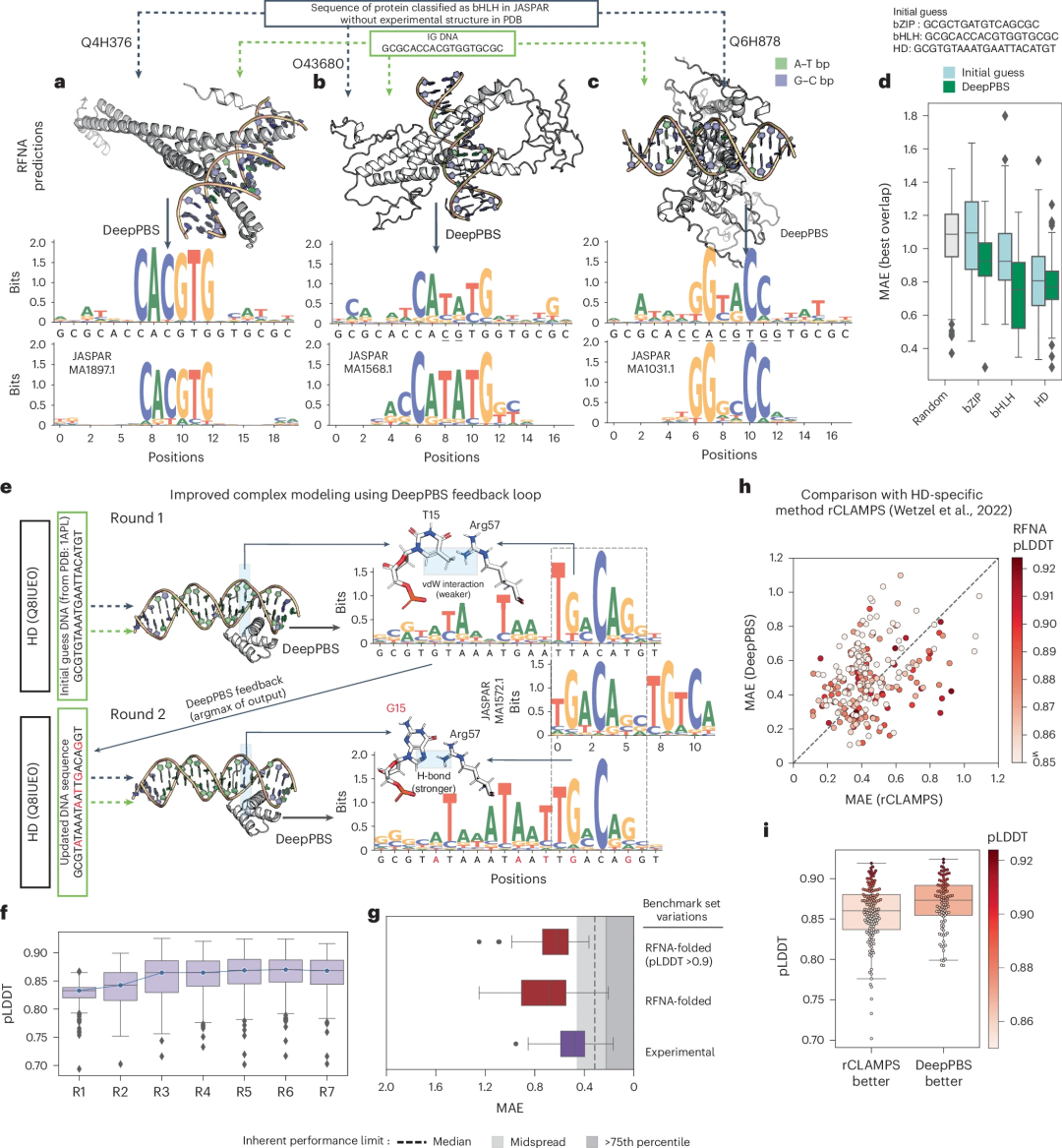
Illustration: Anwendung von DeepPBS bei der Vorhersage der Struktur von Protein-DNA-Komplexen. (Quelle: Papier)
Im Hinblick auf die Interpretierbarkeit können die „relativen Wichtigkeitswerte“ (RI) verschiedener schwerer Atome in Proteinen, die mit DNA interagieren, aus DeepPBS extrahiert werden.
Als Fallstudie zu Proteinen, die für die Krebsentstehung wichtig sind, analysierten die Forscher die p53-DNA-Schnittstelle anhand dieser RI-Scores und verknüpften sie zur Validierung mit vorhandener Literatur.
Und die DeepPBS-Ergebnisse stimmen gut mit dem vorhandenen Wissen überein und können aggregiert werden, um eine angemessene Übereinstimmung mit Alanin-Scanning-Mutagenese-Experimenten zu erzielen.
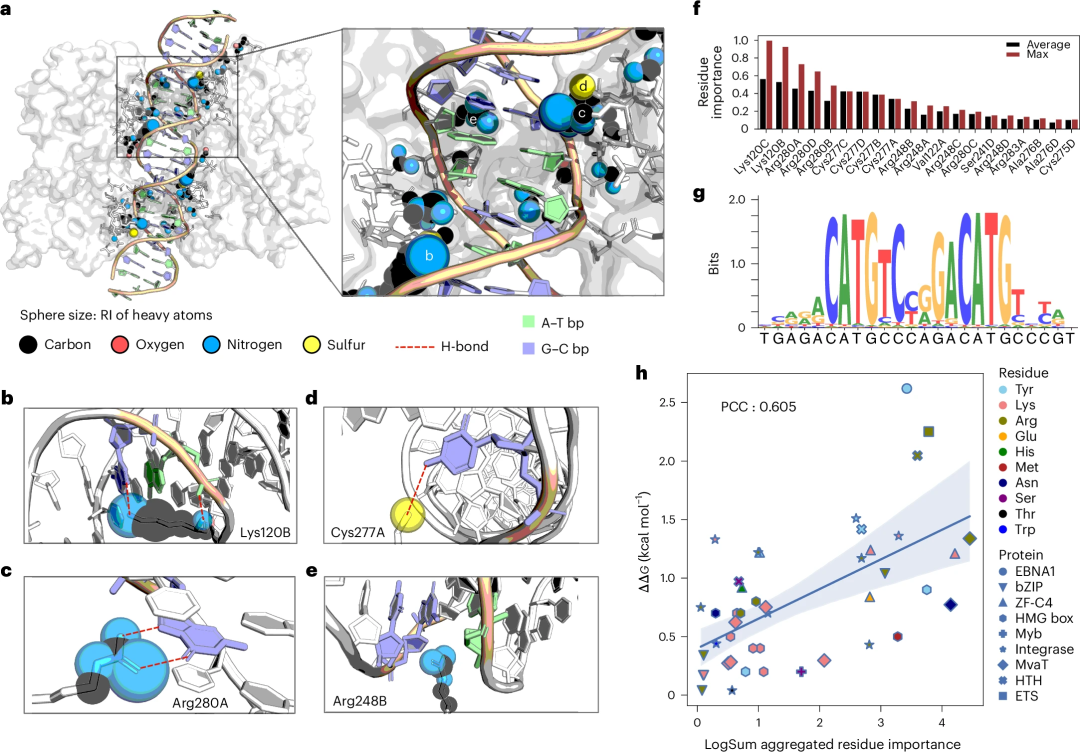
Abbildung: Nehmen wir die Visualisierung der DeepPBS-Wichtigkeitswerte in der p53-DNA-Schnittstelle als Beispiel für die Untersuchung und Durchführung experimenteller Verifizierung. (Quelle: Papier)
In weiteren Proof-of-Principle-Studien wandten Forscher DeepPBS auf in silico entworfene Protein-DNA-Komplexe an, die auf bestimmte DNA-Sequenzen aus einem kürzlich durchgeführten Experiment abzielten, das Strukturdesign und DNA-Mutageneseforschung kombinierte. DeepPBS kann auch zur Analyse molekularer Simulationsverläufe verwendet werden.
„Für Forscher ist es wichtig, eine Methode zu finden, die für alle Proteine funktioniert und nicht auf eine gut untersuchte Proteinfamilie beschränkt ist. Diese Methode ermöglicht es uns auch, neue Proteine zu entwerfen.“
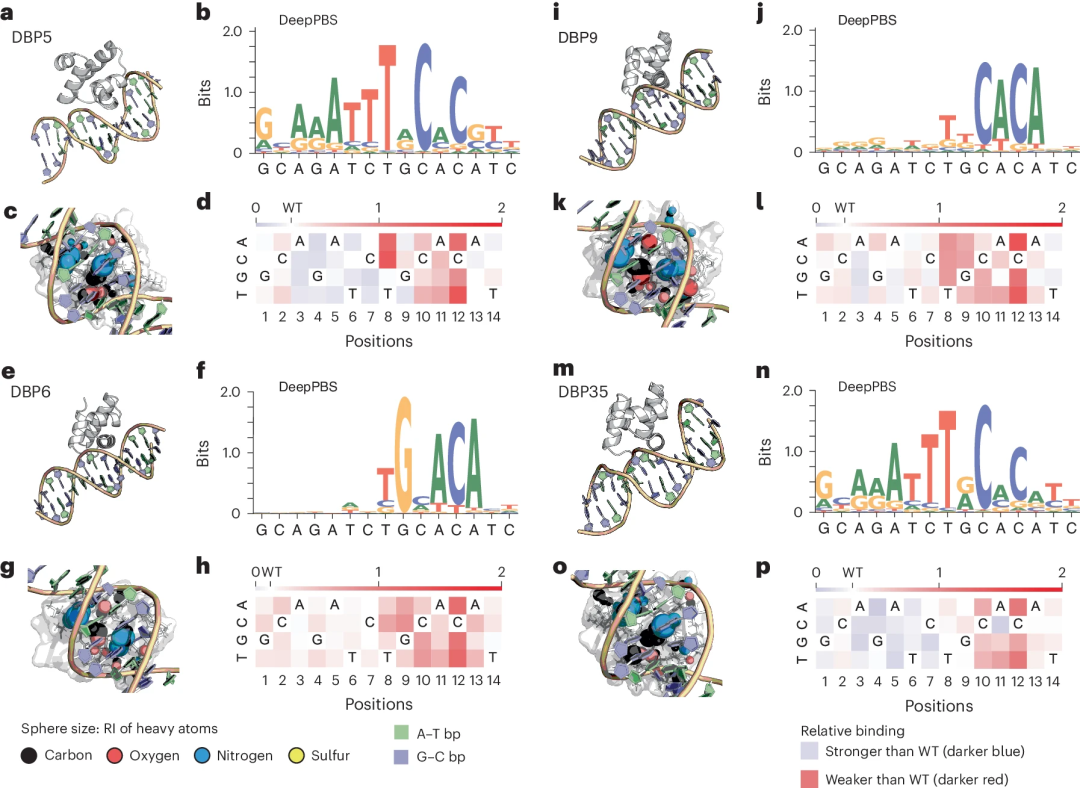
Abbildung: Anwendung von DeepPBS auf in silico entworfene HTH-Gerüste, die auf bestimmte DNA-Sequenzen abzielen. (Quelle: Papier)
Die aktuelle Version von DeepPBS weist inhärente Einschränkungen auf. Es ist auf doppelsträngige DNA zugeschnitten und funktioniert noch nicht mit einzelsträngiger DNA, RNA oder chemisch modifizierten Basen.
Das Modell kann jedoch erweitert werden, um diese unterschiedlichen Szenarien sowie andere Polymer-Polymer-Wechselwirkungen und möglicherweise auch mechanistische Mutationen zu berücksichtigen. Die DeepPBS-Architektur kann im Hinblick auf Anwendungs- und Engineering-Erweiterungen optimiert und erweitert werden.
Trotzdem sagte Rohs, dass DeepPBS ein breites Anwendungsspektrum haben wird. Dieser neue Forschungsansatz könnte die Entwicklung neuer Medikamente und Behandlungen beschleunigen, die auf spezifische Mutationen in Krebszellen abzielen, und zu neuen Entdeckungen in der synthetischen Biologie und Anwendungen in der RNA-Forschung führen.
DeepPBS:https://deeppbs.usc.edu
Das obige ist der detaillierte Inhalt vonUm die Protein-DNA-Bindungsspezifität vorherzusagen, entwickelt das USC-Team eine neue geometrische Deep-Learning-Methode. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
 Anwendung künstlicher Intelligenz im Leben
Anwendung künstlicher Intelligenz im Leben Was ist das Grundkonzept der künstlichen Intelligenz?
Was ist das Grundkonzept der künstlichen Intelligenz? Was soll ich tun, wenn sich mein Computer nicht einschalten lässt?
Was soll ich tun, wenn sich mein Computer nicht einschalten lässt? Methode zur Reparatur von Datenbankzweifeln
Methode zur Reparatur von Datenbankzweifeln Zusammenfassung häufig verwendeter Computer-Tastenkombinationen
Zusammenfassung häufig verwendeter Computer-Tastenkombinationen Aktueller Ripple-Preis
Aktueller Ripple-Preis So verwenden Sie Excel-Makros
So verwenden Sie Excel-Makros Windows ändert den Dateityp
Windows ändert den Dateityp



