

Autor | Shanghai Jiao Tong University, Shanghai Artificial Intelligence Laboratory
Herausgeber | Radiologiescans, gesteuert durch Texteingabeaufforderungen), auf medizinischen 3D-Bildern (CT, MR, PET), basierend auf Texteingabeaufforderungen, um eine universelle Segmentierung von 497 Arten von Organen/Läsionen im menschlichen Körper zu erreichen. Alle Daten, Codes und Modelle sind Open Source.
 Papier-Link:
Papier-Link:
Code-Link:
https://github.com/zhaoziheng/SATDaten-Link:
https://github .com/zhaoziheng/SAT-DS/Forschungshintergrund
Die medizinische Bildsegmentierung spielt eine wichtige Rolle bei einer Reihe klinischer Aufgaben wie Diagnose, Operationsplanung und Krankheitsüberwachung. Allerdings trainiert die traditionelle Forschung „dedizierte“ Modelle für jede spezifische Segmentierungsaufgabe, was dazu führt, dass jedes „dedizierte“ Modell einen relativ begrenzten Anwendungsbereich hat und nicht in der Lage ist, ein breites Spektrum medizinischer Segmentierungsanforderungen effizient und bequem zu erfüllen.
Gleichzeitig haben große Sprachmodelle in letzter Zeit große Erfolge im medizinischen Bereich erzielt, und um die Entwicklung allgemeiner medizinischer künstlicher Intelligenz weiter voranzutreiben, ist es notwendig geworden, ein medizinisches Segmentierungstool zu entwickeln, das Sprach- und Positionierungsfähigkeiten verbinden kann.
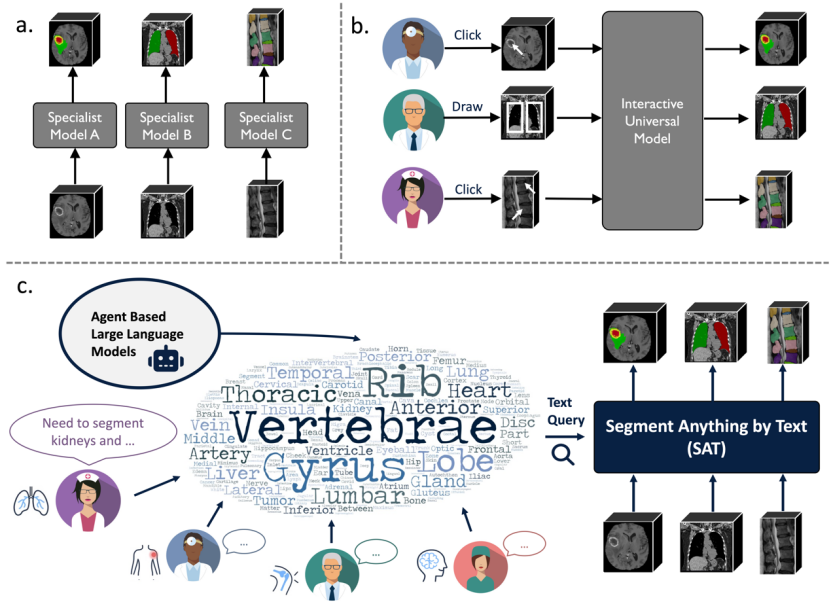 Abbildung 1: SAT unterscheidet sich grundlegend von bestehenden Segmentierungsrahmen.
Abbildung 1: SAT unterscheidet sich grundlegend von bestehenden Segmentierungsrahmen.
1. Diese Studie ist die erste, die die Einspeisung von Wissen über die menschliche Anatomie in einen Textkodierer untersucht, um anatomische Begriffe genau zu kodieren und ein allgemeines medizinisches Segmentierungsmodell für radiologische Bilder zu erstellen .
2. Diese Forschung erstellt den ersten multimodalen medizinischen Wissensgraphen, der mehr als 6.000 Konzepte der menschlichen Anatomie enthält. Gleichzeitig wurde der größte 3D-Datensatz zur medizinischen Bildsegmentierung namens SAT-DS erstellt, der 72 öffentliche Datensätze, mehr als 22.000 Bilder von CT-, MR- und PET-Modalitäten sowie mehr als 302.000 Segmentierungsanmerkungen vereint und den menschlichen Körper abdeckt 497 Segmentierungsziele in 8 Hauptteile.
3. Basierend auf SAT-DS trainierte diese Studie zwei Modelle unterschiedlicher Größe: SAT-Pro (447 Millionen Parameter) und SAT-Nano (110 Millionen Parameter) und entwarf Experimente, um den Wert von SAT aus mehreren Blickwinkeln zu überprüfen: SAT The Die Leistung entspricht der von 72 nnU-Nets-Expertenmodellen (Parameter werden für jeden Datensatz separat angepasst und optimiert, insgesamt etwa 2,2 Milliarden Parameter) und zeigt eine stärkere Generalisierungsfähigkeit für Daten außerhalb der Domäne Als grundlegendes Segmentierungsmodell, das auf großen Datenmengen vorab trainiert wurde, kann SAT bei der Übertragung auf bestimmte Aufgaben durch nachgelagerte Feinabstimmung eine bessere Leistung erzielen Genaue Leistung basierend auf Texteingabeaufforderungen. Schließlich zeigte das Forschungsteam, dass SAT als Proxy-Tool für große Sprachmodelle verwendet werden kann und letztere direkt mit Lokalisierungs- und Segmentierungsfunktionen für Aufgaben wie z als Berichtserstellung.
Im Folgenden werden die Details des Originalartikels aus drei Aspekten vorgestellt: Daten, Modell und experimentelle Ergebnisse.
DatenkonstruktionMultimodaler Wissensgraph:
Um eine genaue Kodierung anatomischer Begriffe zu erreichen, sammelte das Forschungsteam zunächst einen multimodalen Wissensgraphen mit mehr als 6.000 menschlichen Anatomiekonzepten, deren Inhalt aus drei stammt Quellen: 1. Unified Medical Language System (UMLS) ist ein biomedizinisches Wörterbuch, das von der U.S. National Library of Medicine erstellt wurde. Das Forschungsteam extrahierte fast 230.000 biomedizinische Konzepte und Definitionen sowie einen Wissensgraphen, der über 1 Million gegenseitige Beziehungen abdeckt.
2. Maßgebliches Anatomiewissen im Internet. Das Forschungsteam überprüfte 6.502 Konzepte der menschlichen Anatomie und rief mithilfe eines abrufgestützten großen Sprachmodells relevante Informationen aus dem Internet ab. Dabei erhielt es mehr als 6.000 Konzepte und Definitionen sowie eine Wissenskarte, die mehr als 38.000 Beziehungen zwischen anatomischen Strukturen abdeckte.
3. Öffentlicher Segmentierungsdatensatz. Das Forschungsteam sammelte einen umfangreichen öffentlichen 3D-Segmentierungsdatensatz für medizinische Bilder und verknüpfte die segmentierten Bereiche durch anatomische Konzepte (Kategoriebezeichnungen) mit dem Wissen in der oben genannten Textwissensdatenbank, um einen visuellen Wissensvergleich zu ermöglichen.
SAT-DS: Um ein universelles Segmentierungsmodell zu trainieren, erstellte das Forschungsteam SAT-DS, die größte 3D-Segmentierungsdatensammlung für medizinische Bilder auf diesem Gebiet. Insbesondere wurden 72 verschiedene öffentliche Segmentierungsdatensätze gesammelt und organisiert, darunter insgesamt 22.186 3D-Bilder, 302.033 Segmentierungsanmerkungen aus drei Modalitäten: CT, MR und PET sowie 497 Segmentierungen, die 8 Hauptregionen des menschlichen Körpers abdecken ( anatomische Struktur oder Läsion).
Um die Unterschiede zwischen heterogenen Datensätzen zu minimieren, standardisierte das Forschungsteam die Ausrichtung, den Voxelabstand, den Grauwert und andere Bildattribute zwischen verschiedenen Datensätzen und benannte die verschiedenen Datensätze mithilfe einer einheitlichen Segmentierungskategorie des anatomischen Terminologiesystems.

Abbildung 3: SAT-DS ist eine umfangreiche und vielfältige 3D-Segmentierungsdatensammlung für medizinische Bilder, die insgesamt 497 Segmentierungskategorien in 8 Hauptbereichen des menschlichen Körpers abdeckt.
Modellarchitektur
Wissensinjektion: Um einen Prompt-Encoder zu bauen, der anatomische Begriffe genau kodieren kann, injizierte das Forschungsteam zunächst multimodales Anatomiewissen mithilfe von kontrastivem Lernen in den Text-Encoder.
Wie in Abbildung a unten gezeigt, werden anatomische Konzepte verwendet, um multimodales Wissen paarweise zu verbinden, und dann werden ein visueller Encoder und ein Text-Encoder verwendet, um visuelles bzw. Textwissen zu kodieren, und Merkmale werden durch Kontrast erlernt Durch visuelle Ausrichtung Merkmale anatomischer Strukturen mit Textwissen im Raum und der Konstruktion von Beziehungen zwischen anatomischen Strukturen lernen wir eine bessere Kodierung anatomischer Konzepte und dienen als Anhaltspunkte für das Training visueller Segmentierungsmodelle.
Universelle Segmentierung basierend auf Texteingabeaufforderungen: Das Forschungsteam entwarf außerdem ein universelles Segmentierungsmodell-Framework basierend auf Texteingabeaufforderungen, wie in Abbildung b unten dargestellt, einschließlich Textcodierer, visuellem Encoder, visuellem Decoder und Eingabeaufforderungsdecoder.
Angesichts der Tatsache, dass dieselbe anatomische Struktur Unterschiede in verschiedenen Bildern aufweist, verwendet der Cue-Decoder (Abfragedecoder) die vom visuellen Encoder ausgegebenen Bildmerkmale, um anatomische Konzeptmerkmale, dh Segmentierungshinweise, zu verbessern. Schließlich wird das Skalarprodukt zwischen dem Segmentierungshinweis und den vom visuellen Decoder ausgegebenen Merkmalen auf Pixelebene berechnet, um das Ergebnis der Segmentierungsvorhersage zu erhalten.

Modellbewertung
Diese Studie vergleicht SAT mit zwei repräsentativen Methoden, nämlich dem „spezialisierten“ Modell nnU-Nets und dem interaktiven allgemeinen Segmentierungsmodell MedSAM. Die Bewertung umfasst zwei Aspekte: In-Domain-Datensatztests (umfassende Segmentierungsleistung) und Zero-Shot-Out-of-Domain-Datensatztests (centerübergreifende Datenmigrationsfunktionen). Die Bewertungsergebnisse werden aus drei Ebenen integriert: Datensatz, Kategorie und menschliche Körperregion:
Kategorie: Die Segmentierungsergebnisse derselben Kategorie zwischen verschiedenen Datensätzen werden zusammengefasst und gemittelt.
Region: Basierend auf den Kategorieergebnissen sind die Kategorieergebnisse innerhalb desselben menschlichen Anatomiebereichs zusammengefasst und gemittelt;
Datensatz: Traditionelle Segmentierungsmodell-Bewertungsmethode, die Segmentierungsergebnisse innerhalb desselben Datensatzes werden gemittelt
Vergleichsexperiment mit dem dedizierten Modell nnU-Nets
Um das zu maximieren Die Leistung von nnU-Nets wurde in der Studie für jedes einzelne nnU-Nets-Training durchgeführt und mit SAT verglichen. Die spezifischen Einstellungen sind wie folgt:
1 Datensätze in SAT-DS werden zum Testen und Vergleichen verwendet. Für SAT wird die Summe von 72 Trainingssätzen zum Training verwendet und auf 72 Testsätzen für nnU-Nets getestet. Die Ergebnisse von 72 nnU-Nets auf ihren jeweiligen Testsätzen werden als Ganzes zusammengefasst.
2. Im Out-of-Domain-Test wurden 72 Datensätze weiter unterteilt und die Trainingssätze von 49 Datensätzen (genannt SAT-DS-Nano) zum Trainieren von SAT-Nano und Zero-Shot-Test verwendet; Für nnU-Nets werden 49 nnU-Nets verwendet, um 10 Testsätze außerhalb der Domäne zu testen, und die Ergebnisse werden zusammengefasst.

In-Domain-Testergebnisse: Wie aus Tabelle 1 ersichtlich ist, zeigte SAT-Pro im In-Domain-Test eine sehr ähnliche Leistung wie 72 nnU-Nets und übertraf nnU-Nets in mehreren Bereichen. Es ist zu beachten, dass SAT 72 Segmentierungsaufgaben mit nur einem Modell ausführen kann und die Modellgröße viel kleiner ist als die Menge der nnU-Netze (wie in Abbildung c unten dargestellt).
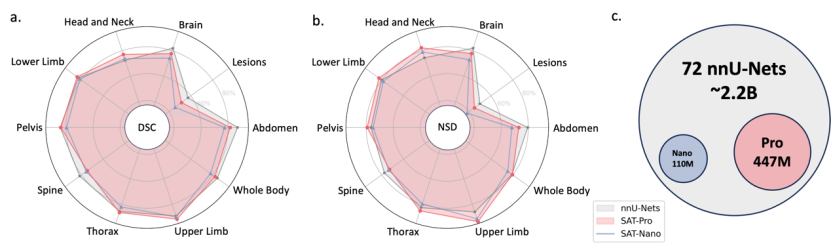
Feinabstimmung der Migrationstestergebnisse: Die Studie testete SAT-Pro für jeden Datensatz nach einer separaten Feinabstimmung weiter, genannt SAT-Pro-Ft. Wie aus Tabelle 1 hervorgeht, weist SAT-Pro-Ft im Vergleich zu SAT-Pro in allen Bereichen erhebliche Leistungsverbesserungen auf und übertrifft nnU-Nets in der Gesamtleistung.
Testergebnisse außerhalb der Domäne: Wie in Tabelle 2 gezeigt, übertraf SAT-Nano nnU-Nets bei 19 der 20 Indikatoren in 10 Datensätzen, was insgesamt stärkere Migrationsfähigkeiten zeigt.
Tabelle 2: Vergleich von Tests außerhalb der Domäne zwischen SAT-Nano, nnU-Nets und MedSAM. Die Ergebnisse werden in Einheiten von Datensätzen dargestellt.

Vergleichsexperiment mit dem interaktiven Segmentierungsmodell MedSAM
Diese Studie verwendet direkt den öffentlichen Prüfpunkt von MedSAM zum Testen und SAT-Vergleich. Die spezifischen Einstellungen sind wie folgt:
1. Im domäneninternen Test, ab 72 Daten Wir haben außerdem 32 Datensätze untersucht, die im MedSAM-Training zum Vergleich verwendet wurden.
2. Im Out-of-Domain-Test wurden 5 Datensätze, die nicht im MedSAM-Training verwendet wurden, zum Vergleich gescreent.
Berücksichtigen Sie für MedSAM zwei verschiedene Box-Eingabeaufforderungen: Verwenden Sie das kleinste Rechteck (Oracle Box), das die Ground-Truth-Segmentierung enthält und als MedSAM (Tight) aufgezeichnet wird. Fügen Sie zufällige Offsets basierend auf der Oracle Box hinzu, die als MedSAM (Loose) aufgezeichnet wird. Testen Sie gleichzeitig die Wirkung von Oracle Box direkt als Prognose. Für SAT wird das Modell im nnU-Nets-Vergleichsexperiment direkt zum Testen dieser Datensätze ohne erneutes Training verwendet.
In-Domain-Testergebnisse:Wie in Tabelle 3 gezeigt, schneidet SAT-Pro in fast allen Bereichen besser ab als MedSAM, und die Gesamtleistung von SAT-Pro und SAT-Nano ist besser als MedSAM. Obwohl SAT-Pro bei Läsionen schlechter abschneidet als MedSAM, schneidet Oracle Box selbst bei Läsionen als Vorhersage gut ab und übertrifft sogar MedSAM bei DSC. Dies weist darauf hin, dass die überlegene Leistung von MedSAM bei der Segmentierung von Läsionen wahrscheinlich auf den starken Vorinformationen von Box beruht.
Tabelle 3: Vergleich der domäneninternen Tests von SAT-Pro, SAT-Nano und MedSAM, die Ergebnisse sind in Einheiten von Regionen oder Läsionen integriert.

Qualitativer Vergleich: Abbildung 6 wählt zwei typische Beispiele aus den Ergebnissen des domäneninternen Tests zur visuellen Darstellung aus, um SAT und MedSAM weiter zu vergleichen. Wie in Abbildung 6 gezeigt, ist es bei der Segmentierung des Myokards schwierig, mit der Box-Eingabeaufforderung zwischen dem Myokard und den vom Myokard umhüllten Ventrikeln zu unterscheiden. Daher hat MedSAM die beiden auch fälschlicherweise zusammen segmentiert, was zeigt, dass die Box-Eingabeaufforderung ähnlich ist komplexe räumliche Beziehungen Es kann leicht zu Unklarheiten kommen, die zu einer ungenauen Segmentierung führen.
Im Gegensatz dazu kann SAT, das auf Texteingabeaufforderungen basiert (direkte Eingabe der Namen anatomischer Strukturen), genau zwischen Myokard und Ventrikeln unterscheiden. Darüber hinaus ist Oracle Box, wie in der in Abbildung 6 gezeigten Darmtumorsegmentierung zu sehen ist, bereits ein gutes Vorhersageergebnis für das Läsionsziel, während das Segmentierungsergebnis von MedSAM möglicherweise nicht besser ist als die erhaltene Box-Eingabeaufforderung.
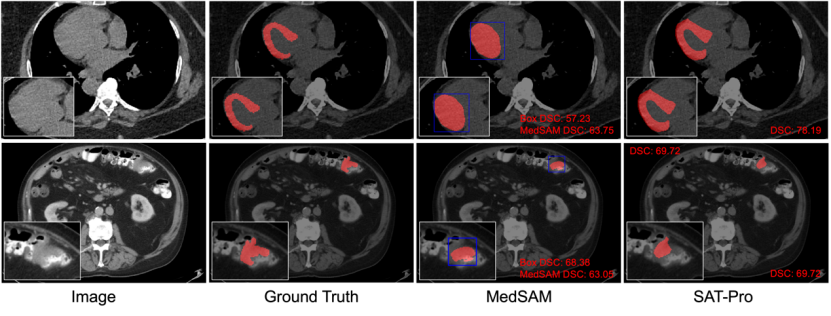
Abbildung 6: Qualitativer Vergleich zwischen SAT-Pro und MedSAM (Tight). Unter anderem verwendet MedSAM Oracle Box als Eingabeaufforderung, und die Box ist blau markiert. Die erste Reihe zeigt ein Beispiel einer Myokardsegmentierung; die zweite Reihe zeigt ein Beispiel einer intestinalen Tumorsegmentierung.
Testergebnisse außerhalb der Domäne: Wie in Tabelle 2 gezeigt, übertraf SAT-Nano MedSAM im Vergleich zu MedSAM (Tight) in 5 von 10 Indikatoren in 5 Datensätzen. MedSAM (Loose) weist bei allen Indikatoren offensichtliche Leistungseinbußen auf, was darauf hindeutet, dass MedSAM empfindlicher auf den vom Benutzer eingegebenen Offset der Box-Eingabeaufforderung reagiert.
Ablationsexperiment
Beim Entwurf von SAT sind das visuelle Backbone-Netzwerk und der Text-Encoder zwei Schlüsselkomponenten. Diese Forschung versucht, verschiedene visuelle Netzwerkstrukturen oder Text-Encoder im SAT-Framework zu verwenden und deren Einfluss durch allgemeine Ablationsexperimente zu untersuchen.
Um Kosten für Experimente zu sparen, werden alle SAT-Modellschulungen und Tests in Ablationsexperimenten mit SAT-DS-Nano durchgeführt, das 49 Datensätze enthält, die 13303 3D-Bilder, 151461 Segmentierungsanmerkungen und 429 Split-Kategorien enthalten.
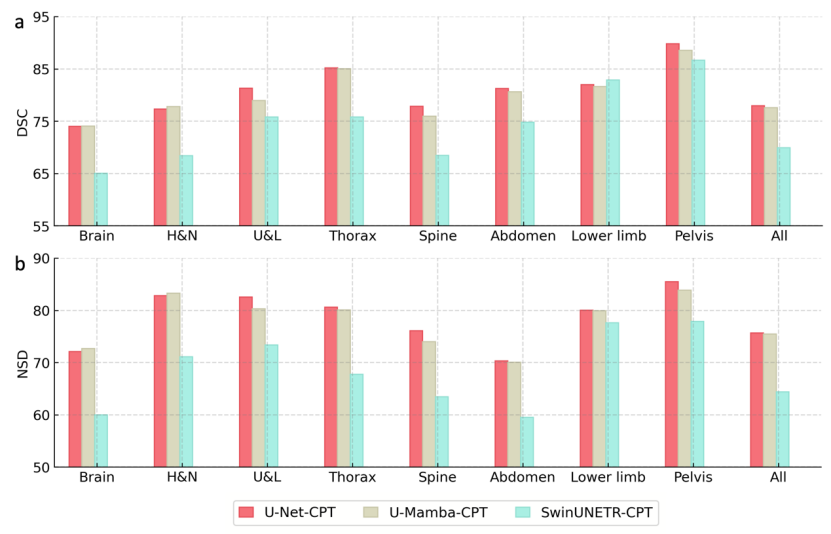
Visuelles Backbone-Netzwerk: Im Rahmen von SAT-Nano wählte diese Studie drei gängige Segmentierungsnetzwerkstrukturen zum Vergleich aus, nämlich U-Net (110 Millionen Parameter), SwinUNETR (107 Millionen Parameter) und U-Mamba (114 Millionen Parameter). Um einen fairen Vergleich zu ermöglichen, sind die Parameterbeträge, die sie in diesem Ablationsexperiment steuern, ungefähr ähnlich. Gleichzeitig wird zur Berechnung des Overheads der Schritt der Wissensinjektion weggelassen und MedCPT direkt verwendet (MedCPT ist ein Textencoder, der auf PubMed-Literatur basiert, mit 225 Millionen Klickdaten privater Benutzer trainiert wurde und die beste Leistung erzielt hat eine Reihe medizinischer Sprachaufgaben), während der Text-Encoder Hinweise generiert. Die drei Varianten werden als U-Net-CPT, SwinUNETR-CPT bzw. U-Mamba-CPT bezeichnet.
Wie Sie in Abbildung 7 sehen können, ist die endgültige Segmentierungsleistung bei Verwendung von U-Net und U-Mamba als visuelles Backbone-Netzwerk relativ ähnlich, wobei U-Net etwas besser ist als U-Mamba, während die Segmentierungsleistung bei Verwendung von SwinUNETR ist ein deutlich besserer Rückgang. Schließlich wählte das Forschungsteam U-Net als visuelles Backbone-Netzwerk für SAT.
Text-Encoder: Im Rahmen von SAT-Nano wurden in dieser Studie drei repräsentative Text-Encoder zum Vergleich ausgewählt: ein Text-Encoder, der mit der oben vorgeschlagenen Wissensinjektionsmethode trainiert wurde (bezeichnet als „unsere“), der Stand der Technik Es wird der medizinische Text-Encoder MedCPT und der nicht für medizinische Daten optimierte Text-Encoder BERT-base verwendet.
Der Fairness halber verwendet dieses Ablationsexperiment einheitlich U-Net als visuelles Netzwerk. Die drei Varianten werden als U-Net-Ours, U-Net-CPT bzw. U-Net-BB bezeichnet. Wie in Abbildung 8 dargestellt, weist die Verwendung von MedCPT im Vergleich zur Verwendung von BERT-Base insgesamt eine leichte Verbesserung der Segmentierungsleistung auf, was darauf hinweist, dass Domänenkenntnisse hilfreich sind, um gute Segmentierungstipps bereitzustellen, während die Verwendung des in dieser Studie vorgeschlagenen Textencoders die beste Leistung erbracht hat in allen Kategorien erreicht, was darauf hindeutet, dass der Aufbau einer multimodalen Wissensbasis zur menschlichen Anatomie und die Wissensinjektion für Segmentierungsmodelle erheblich hilfreich sind.

Die Long-Tail-Verteilung ist ein offensichtliches Merkmal segmentierter Datensätze. Wie in Abbildung 9 a und b dargestellt, untersuchte das Forschungsteam die Verteilung der Anzahl der Anmerkungen von 429 Kategorien in SAT-DS-Nano, die für Ablationsexperimente verwendet wurden. Wenn die 10 Kategorien mit der größten Anzahl von Anmerkungen (oberste 2,33 %) als Kopfklassen definiert werden und die 150 Kategorien mit der geringsten Anzahl von Anmerkungen (letzte 34,97 %) als Endklassen definiert werden, kann die Anzahl ermittelt werden Anmerkungen für Tail-Klassen machen nur 3,25 % der Gesamtzahl der Anmerkungen aus.
Diese Studie untersucht weiter den Einfluss von Textkodierern auf die Segmentierungsergebnisse verschiedener Kategorien in Long-Tail-Verteilungen. Wie in Abbildung 9c dargestellt, erzielte der vom Forschungsteam vorgeschlagene Encoder die beste Leistung in den Kategorien Kopf, Schwanz und Mitte, wobei die Verbesserung in der Kategorie Schwanz deutlicher war als in der Kategorie Kopf. Gleichzeitig schneidet MedCPT in der Kopfklasse etwas schlechter ab als BERT-Basis, in der Schwanzklasse jedoch besser. Diese Ergebnisse zeigen, dass Domänenwissen, insbesondere die Einbeziehung von multimodalem Wissen über die menschliche Anatomie, für die Segmentierung von Long-Tail-Kategorien erheblich hilfreich ist.

Kombiniert mit großen Sprachmodellen
Da SAT basierend auf Textaufforderungen segmentiert werden kann, kann es direkt als Proxy-Tool für große Sprachmodelle verwendet werden, um Segmentierungsfunktionen bereitzustellen. Um Anwendungsszenarien zu demonstrieren, wählte das Forschungsteam vier verschiedene reale klinische Daten aus, extrahierte mithilfe von GPT4 Segmentierungsziele aus dem Bericht und rief SAT für die Zero-Shot-Segmentierung auf. Die Ergebnisse sind in Abbildung 10 dargestellt.
Wie Sie sehen, kann GPT-4 die wichtigen anatomischen Strukturen im Bericht sehr gut erkennen und SAT aufrufen, um diese sehr gut auf echten klinischen Bildern zu segmentieren, ohne dass eine Feinabstimmung der Daten erforderlich ist.

Forschungswert
Als erstes groß angelegtes allgemeines Segmentierungsmodell für medizinische 3D-Bilder basierend auf Textaufforderungen spiegelt sich der Wert von SAT in vielen Aspekten wider:
SAT erstellt eine effiziente und flexible universelle Segmentierung: SAT-Pro verwendet nur ein Modell, zeigt bei einem breiten Spektrum von Segmentierungsaufgaben eine vergleichbare Leistung wie 72 nnU-Nets und verfügt über eine geringere Anzahl an Modellparametern. Dies zeigt, dass SAT-Pro als allgemeines Segmentierungsmodell im Vergleich zu herkömmlichen medizinischen Segmentierungsmethoden, die die Konfiguration, Schulung und Bereitstellung einer Reihe spezialisierter Modelle erfordern, eine flexiblere und effizientere Lösung ist. Gleichzeitig bewies das Forschungsteam auch, dass SAT-Pro eine bessere Generalisierungsleistung bei Daten außerhalb der Region aufweist und klinische Anforderungen wie die zentrumsübergreifende Migration besser erfüllen kann.
SAT ist ein Basismodell, das auf dem Vortraining großer Segmentierungsdaten basiert: Nachdem SAT-Pro an einem großen Segmentierungsdatensatz trainiert wurde, zeigt es erhebliche Leistungsverbesserungen, wenn es durch Feinabstimmung auf einen bestimmten Datensatz übertragen wird. Tuning und bietet insgesamt eine bessere Leistung als nnU-Nets. Dies weist darauf hin, dass SAT als leistungsstarkes Basissegmentierungsmodell angesehen werden kann, das durch eine fein abgestimmte Übertragung eine bessere Leistung bei bestimmten Aufgaben erbringen kann und so die klinischen Anforderungen der Allzwecksegmentierung und der Spezialsegmentierung in Einklang bringt.
SAT erreicht eine genaue und robuste Segmentierung basierend auf Texteingabeaufforderungen: Im Vergleich zum interaktiven Segmentierungsmodell basierend auf Box-Eingabeaufforderungen kann SAT basierend auf Texteingabeaufforderungen genauere und Eingabeaufforderungs-robustere Segmentierungsergebnisse erzielen und Benutzern viel Zeit ersparen Zeitaufwand für das Zeichnen von Boxen, wodurch eine automatische und stapelweise universelle Segmentierung erreicht wird.
SAT kann als Proxy-Tool für große Sprachmodelle verwendet werden: Das Forschungsteam zeigte anhand realer klinischer Daten, dass SAT nahtlos mit großen Sprachmodellen verbunden werden kann und Text als Brücke nutzt, um direkt Segmentierungs- und Positionierungsmöglichkeiten für alle bereitzustellen großes Sprachmodell. Dies ist von großem Wert, um die Entwicklung der generalistischen medizinischen künstlichen Intelligenz weiter voranzutreiben.
Der Einfluss der Modellgröße auf die Segmentierung: Durch das Training von zwei Modellen unterschiedlicher Größe: SAT-Nano und SAT-Pro konnte in dieser Studie beobachtet werden, dass SAT-Pro im Vergleich zu SAT-Nano im domäneninternen Test eine deutliche Verbesserung aufweist . Dies impliziert, dass das Skalierungsgesetz weiterhin gilt, wenn allgemeine Segmentierungsmodelle für große Datensätze trainiert werden.
Der Einfluss von Domänenwissen auf die Segmentierung: Das Forschungsteam schlug die erste multimodale Wissensdatenbank zur menschlichen Anatomie vor und untersuchte den Einsatz von Wissenserweiterung zur Verbesserung der Leistung allgemeiner Segmentierungsmodelle, insbesondere der Segmentierung von Long-Tail-Kategorien. Angesichts der Tatsache, dass Segmentierungsanmerkungen, insbesondere Anmerkungen zu Long-Tail-Kategorien, relativ selten sind, ist diese Untersuchung für die Erstellung eines allgemeinen Segmentierungsmodells von großer Bedeutung.
Das obige ist der detaillierte Inhalt vonDas Open-Source-3D-Medizin-Großmodell SAT unterstützt 497 Organoide und hat eine Leistung von über 72 nnU-Nets. Es wurde vom Team der Shanghai Jiao Tong University veröffentlicht.. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
 besonderer Symbolpunkt
besonderer Symbolpunkt
 file_get_contents
file_get_contents
 So entfernen Sie Personen von der Blacklist auf WeChat
So entfernen Sie Personen von der Blacklist auf WeChat
 So importieren Sie MDF-Dateien in die Datenbank
So importieren Sie MDF-Dateien in die Datenbank
 Der Geschwindigkeitsunterschied zwischen USB2.0 und 3.0
Der Geschwindigkeitsunterschied zwischen USB2.0 und 3.0
 Welche Rolle spielt die Kafka-Verbrauchergruppe?
Welche Rolle spielt die Kafka-Verbrauchergruppe?
 So beheben Sie den HTTP-Fehler 503
So beheben Sie den HTTP-Fehler 503
 Warum kann Swoole im Speicher verbleiben?
Warum kann Swoole im Speicher verbleiben?




